Secuenciación transcriptómica de células individuales a partir de ADNc 3′ preparado con 10x Genomics y los kits SQK-LSK114 y EXP-PCA001 (SST_9198_v114_revP_06Oct2025)
PromethION: Protocol
Secuenciación transcriptómica de células individuales a partir de ADNc 3′ preparado con 10x Genomics y los kits SQK-LSK114 y EXP-PCA001 V SST_9198_v114_revP_06Oct2025
Método de transcriptómica de células individuales:
- Requiere amplicones de ADNc generados mediante los ensayos Chromium GEM-X Universal 3’ Gene Expression (V4) o Chromium Next GEM Universal 3’ Gene Expression (V3.1) de 10x Genomics.
- Tiempo de preparación de la biblioteca ~210 min
- Gran rendimiento
- Requiere PCR
De uso exclusivo en investigación
FOR RESEARCH USE ONLY
Contents
Introducción al protocolo
Preparación de la biblioteca
- 3. PCR previa a la captura
- 4. Captura
- 5. PCR posterior a la captura
- 6. Preparación de extremos
- 7. Ligación de los adaptadores y purificación
- 8. Acondicionamiento y carga de celdas de flujo PromethION
Secuenciación y análisis de datos
- 9. Adquisición de datos e identificación de bases
- 10. Reutilización y retorno de celdas de flujo
- 11. Análisis de datos
Resolución de problemas
Descripción general
Método de transcriptómica de células individuales:
- Requiere amplicones de ADNc generados mediante los ensayos Chromium GEM-X Universal 3’ Gene Expression (V4) o Chromium Next GEM Universal 3’ Gene Expression (V3.1) de 10x Genomics.
- Tiempo de preparación de la biblioteca ~210 min
- Gran rendimiento
- Requiere PCR
De uso exclusivo en investigación
1. Introducción
Introducción al protocolo de transcriptómica 3’ de células individuales
Esta aplicación permite secuenciar transcritos completos de ADNc 3’, lo que proporciona una visión integral de las isoformas expresadas y permite detectar eventos de empalme alternativo y fusiones en células individuales. Además, permite detectar SNP en cualquier parte del transcrito, así como identificar subtipos celulares en una población en función de los niveles de expresión de las isoformas.
Este protocolo describe cómo llevar a cabo la secuenciación de ADNc a partir de células individuales con el kit Ligation Sequencing V14 (SQK-LSK114) y la mezcla de amplificación por PCR (EXP-PCA001). Se deberá haber retrotranscrito el ARNm obtenido de células individuales en ADNc mediante los ensayos Chromium GEM-X Universal 3’ Gene Expression (V4) o Chromium Next GEM Universal 3’ Gene Expression (V3.1), de 10x Genomics. A continuación, se debe marcar el ADNc con biotina antes de la amplificación por PCR con oligos sintetizados por encargo. Después, se realiza la captura de los amplicones en microesferas de streptavidina, antes de una segunda PCR con los cebadores de PCR (PRM). Por último, se realiza una preparación de biblioteca normal del kit de secuenciación por ligación V14, con la que se preparan los extremos de ADNc para secuenciar en un dispositivo PromethION.
Este protocolo se ha optimizado a partir del método High throughput error corrected Nanopore single cell transcriptome sequencing (Nat Commun 11, 4025 (2020), descrito por Lebrigand, K., Magnone, V., Barbry, P. et al. y se utiliza para secuenciar transcritos completos, eliminar artefactos de síntesis de ADNc y corregir el sesgo de hebra.
Nota: este protocolo está pensado para utilizarse con los ensayos Chromium GEM-X Universal 3’ Gene Expression (V4) o Chromium Next GEM Universal 3’ Gene Expression (V3.1) de 10x Genomics.
Si utiliza los ensayos Chromium GEM-X Universal 5' Gene Expression (V3) y Chromium Next GEM Universal 5' Gene Expression (V2) de 10x Genomics, consulte el protocolo Single-cell transcriptomics sequencing from 5’ cDNA prepared with 10x Genomics using SQK-LSK114.
Si utiliza los ensayos Visium Spatial Gene Expression (Visium V1 para muestras congeladas en fresco) o Visium HD 3' Spatial Gene Expression, consulte el protocolo Spatial transcriptomics sequencing from 3’ cDNA prepared with 10x Genomics using SQK-LSK114 and EXP-PCA001.
Pasos del proceso de secuenciación:
Preparación del experimento
Pasos:
- Prepare con anterioridad ADNc con códigos de barras de células individuales, mediante los ensayos Chromium GEM-X Universal 3’ Gene Expression (V4) o Chromium Next GEM Universal 3’ Gene Expression (V3.1) de 10x Genomics. Utilice una muestra inicial de buena calidad. Los controles de calidad realizados durante el protocolo son esenciales para garantizar el éxito del experimento.
- Tener a disposición el kit de secuenciación, el instrumental pertinente y los reactivos de otros fabricantes.
- Descargar el programa MinKNOW para obtener y analizar los datos.
- Comprobar la(s) celda(s) de flujo para asegurarse de que tiene(n) poros suficientes para una buena secuenciación.
Preparación de la biblioteca
| Paso del protocolo | Proceso | Duración | Parada opcional |
|---|---|---|---|
| PCR previa a la captura | Marcar el ADNc con biotina y amplificar por PCR | 60 min | - |
| Captura | Capturar los amplicones mediante microesferas con streptavidina | 40 min | - |
| PCR posterior a la captura | Amplificar los amplicones por PCR con cebadores de PCR (PRM) | 50 min | - |
| Preparación de extremos | Preparar los extremos de ADNc para ligar el adaptador | 30 min | a 4 °C durante la noche |
| Ligación de los adaptadores y purificación | Ligar los adaptadores de secuenciación al ADNc | 20 min | a 4 °C en almacenamiento a corto plazo o durante un uso repetido, como para las recargas de la celda de flujo a –80 °C en almacenamiento a largo plazo |
| Acondicionar y cargar la celda de flujo | Acondicionar la celda de flujo y cargar la biblioteca | 10 min | |
Secuenciación y análisis
Pasos:
- Empezar el proceso de secuenciación desde el programa MinKNOW, que obtendrá datos brutos del dispositivo e identificará las lecturas.
- Analizar los datos con el flujo bioinformático wf-single-cell de EPI2ME.
Compatibilidad del protocolo
Este protocolo debería usarse solo junto con los siguientes productos:
- Kit de secuenciación por ligación V14 (SQK-LSK114)
- Material de amplificación PCR Expansion (EXP-PCA001)
- Celdas de flujo R10.4.1 (FLO-PRO114M)
- Kit Flow Cell Wash (EXP-WSH004)
- Dispositivo PromethION - Requisitos informáticos PromethION 24/48
2. Material y consumibles
Material
- 10 ng de amplicones de ADNc producidos con los ensayos Chromium GEM-X Universal 3’ Gene Expression (V4) o Chromium Next GEM Universal 3’ Gene Expression (V3.1) de 10x Genomics
- Oligo de encargo a 10 μM: [Btn]Fwd_3580_partial_read1_defined_for_3'_cDNA (secuencia indicada a continuación)
- Oligo de encargo a 10 μM: Rev_PR2_partial_TSO_defined_for_3'_cDNA (secuencia indicada a continuación)
- Kit Ligation Sequencing V14 (SQK-LSK114)
- PCR Expansion (EXP-PCA001)
Consumibles
- Celdas de flujo PromethION (FLO-PRO114M)
- LongAmp Hot Start Taq 2X Master Mix (NEB, M0533)
- Módulo NEBNext® Ultra™ II End Repair/dA-Tailing (NEB, E7546)
- Ligasa de ADN Salt-T4® (NEB, M0467)
- Kit Qubit 1x dsDNA HS (ThermoFisher, Q33230)
- Kit Agilent Technologies DNA 12000
- Streptavidina M280, 10 μg/μl (Invitrogen, 11205D)
- Microesferas Agencourt AMPure XP (Beckman Coulter™, A63881)
- Tris-HCl 1 M, pH 7,5
- NaCl 5 M (Sigma, 71386)
- EDTA 0,5 M, pH 8 (Thermo Scientific, R1021)
- Etanol al 80 % recién preparado con agua sin nucleasas
- Nuclease-free water (e.g. Thermo Scientific, AM9937)
- Tubos Falcon de 15 ml
- 1.5 ml Eppendorf DNA LoBind tubes
- 0.2 ml thin-walled PCR tubes
- Qubit™ Assay Tubes (Invitrogen, Q32856)
Instrumental
- Pantalla protectora celdas de flujo PromethION
- Dispositivo PromethION
- Bioanalizador Agilent (o equivalente)
- Hula mixer (gentle rotator mixer)
- Gradilla magnética (p. ej., Invitrogen DynaMag-2 Magnet, 12321D)
- Microfuge
- Vortex mixer
- Thermal cycler
- P1000 pipette and tips
- P200 pipette and tips
- P100 pipette and tips
- P20 pipette and tips
- P10 pipette and tips
- P2 pipette and tips
- Ice bucket with ice
- Timer
- Qubit™ fluorometer (or equivalent for QC check)
En este protocolo, se necesitan 10 ng de amplicones de ADNc amplificados, preparados con uno de los ensayos Chromium GEM-X Universal 3’ Gene Expression (V4) o Chromium Next GEM Universal 3’ Gene Expression (V3.1) de 10x Genomics.
Kits de 10x Genomics
Nota: este protocolo está pensado para utilizarse con los 10 ng de amplicones de ADNc preparados mediante uno de los ensayos de 10x Genomics mencionados anteriormente: Chromium GEM-X Universal 3’ Gene Expression (V4) o Chromium Next GEM Universal 3’ Gene Expression (V3.1).
El protocolo Single-cell transcriptomics sequencing from 5’ cDNA prepared with 10x Genomics using SQK-LSK114 está pensado para utilizarse con uno de los ensayos Chromium GEM-X Universal 5' Gene Expression (V3) o Chromium Next GEM Universal 5' Gene Expression (V2) de 10x Genomics.
Si utiliza los ensayos Visium Spatial Gene Expression (Visium V1 para muestras congeladas en fresco) o Visium HD 3' Spatial Gene Expression, consulte el protocolo Spatial transcriptomics sequencing from 3’ cDNA prepared with 10x Genomics using SQK-LSK114 and EXP-PCA001.
Secuencias de oligonucleótidos personalizadas
Pida los siguientes oligonucleótidos purificados por HPLC a una concentración de 100 μM y diluya a 10 μM en tampón TE para utilizarlos en el paso previo a la captura de la preparación de la biblioteca.
| Nombre | Secuencia |
|---|---|
| [Btn]Fwd_3580_partial_read1_defined_for_3'_cDNA | 5'-/5Biosg/CAGCACTTGCCTGT CGCTCTATCTTCCTACA CGACGCTCTTCCGATCT-3' |
| Rev_PR2_partial_TSO_defined_for_3'_cDNA | 5'-CAGCTTTCTGTTGGTGCTGA TATTGCAAGCAGTGGTA TCAACGCAGAG-3' |
Microesferas AMPure XP
En los primeros pasos, cuando se preparen los amplicones de ADNc se necesitará una cantidad adicional de microesferas AMPure XP. Desde el paso de preparación de extremos, es posible utilizar las microesferas AMPure XP del kit Ligation Sequencing V14 (SQK-LSK114).
Muestra inicial de ADN
Verificar la calidad de la muestra inicial
Es importante que la muestra inicial cumpla con los requisitos de cantidad y calidad. Usar demasiado ADN, poco o de mala calidad (p. ej., que esté muy fragmentado, que contenga ARN o contaminantes químicos), tal vez afecte a la preparación de la biblioteca.
Siga las instrucciones sobre cómo realizar un control de calidad de la muestra de ADN, en el protocolo Input DNA/ RNA QC.
Contaminantes químicos
Dependiendo de cómo se extraiga el ADN de la muestra original, es posible que ciertos contaminantes químicos permanezcan en el ADN purificado, lo cual tal vez afecte la eficacia de la preparación de la biblioteca y la calidad de la secuenciación. Encontrará más información sobre contaminantes en la página Contaminants de la comunidad Nanopore.
Reactivos de otros fabricantes
Hemos probado y recomendamos el uso de todos los reactivos de otros fabricantes usados en este protocolo. Oxford Nanopore Technologies no ha evaluado otras alternativas.
Recomendamos preparar estos reactivos siguiendo las instrucciones del fabricante.
Evaluación de la celda de flujo
Antes de empezar el experimento de secuenciación, recomendamos comprobar el número de poros disponibles, presentes en la celda de flujo. La evaluación de la celda de flujo deberá realizarse dentro de las 12 semanas posteriores a la compra de sus celdas de flujo PromethION. Oxford Nanopore Technologies reemplazará cualquier celda de flujo sin usar que tenga menos poros que los indicados en la tabla a continuación, siempre que el resultado se comunique dentro de los dos días posteriores a la realización de la evaluación de la celda de flujo y se hayan seguido las recomendaciones de almacenamiento. Para evaluar la celda de flujo, consultar las instrucciones en el documento Flow Cell Check.
| Celdas de flujo | Cantidad mínima de poros activos cubierta por la garantía |
|---|---|
| PromethION | 5000 |
Contenido del material PCR Expansion (EXP-PCA001)
Nota: en este protocolo, solo se necesitan cebadores de PCR (PRM).
Contenido del kit Ligation Sequencing Kit V14 (SQK-LSK114)
Notas:
- Este producto contiene reactivo AMPure XP, fabricado por Beckman Coulter, Inc., que es posible conservar junto con el kit, a –20 °C, sin perjudicar su estabilidad.
- DNA Control Sample (DCS) es un amplicón estándar de 3,6 kb que se alinea con el extremo 3’ del genoma Lambda.
3. PCR previa a la captura
Material
- 10 ng de amplicones de ADNc producidos con los ensayos Chromium GEM-X Universal 3’ Gene Expression (V4) o Chromium Next GEM Universal 3’ Gene Expression (V3.1) de 10x Genomics
- Oligo de encargo a 10 μM: [Btn]Fwd_3580_partial_read1_defined_for_3'_cDNA (secuencia indicada en la sección Material y consumibles)
- Oligo de encargo a 10 μM: Rev_PR2_partial_TSO_defined_for_3'_cDNA (secuencia indicada en la sección Material y consumibles)
Consumibles
- LongAmp Hot Start Taq 2X Master Mix (NEB, M0533)
- Microesferas Agencourt AMPure XP (Beckman Coulter™, A63881)
- Etanol al 80 % recién preparado con agua sin nucleasas
- Agua sin nucleasas (p. ej., ThermoFisher, AM9937)
- 1.5 ml Eppendorf DNA LoBind tubes
- 0.2 ml thin-walled PCR tubes
Instrumental
- Thermal cycler
- Microfuge
- Hula mixer (gentle rotator mixer)
- Gradilla magnética (p. ej., Invitrogen DynaMag-2 Magnet, 12321D)
- Ice bucket with ice
- P1000 pipette and tips
- P200 pipette and tips
- P100 pipette and tips
- P20 pipette and tips
- P2 pipette and tips
Evaluación de la celda de flujo.
Recomendamos verificar la celda de flujo antes de empezar a preparar la biblioteca y comprobar que tiene poros suficientes para realizar una buena secuenciación.
Para más información, consultar las instrucciones de comprobación de la celda de flujo en el protocolo de MinKNOW.
Preparar los amplicones de ADNc en agua sin nucleasas:
- Transferir 10 ng de amplicones de ADNc a un tubo de PCR de pared fina (0,2 ml).
- Ajustar el volumen a un total de 21 µl con agua sin nucleasas.
- Mezclar golpeando suavemente el tubo con el dedo para evitar fragmentaciones indeseadas.
- Centrifugar brevemente en una microcentrífuga.
En el mismo tubo de PCR de pared fina (0,2 ml), preparar la siguiente reacción de marcado con biotina:
| Reactivo | Concentración inicial | Concentración final | Volumen |
|---|---|---|---|
| Molde de ADNc | 0,48 ng/μl | 0,2 ng/μl | 21 μl |
| [Btn]Fwd_3580_partial_read1_defined_for_3'_cDNA | 10 μM | 0,4 μM | 2 μl |
| Rev_PR2_partial_TSO_defined_for_3'_cDNA | 10 μM | 0,4 μM | 2 μl |
| LongAmp Hot Start Taq 2X Master Mix | 2× | 1× | 25 μl |
| Total | - | - | 50 μl |
Mezclar con la pipeta y centrifugar brevemente.
Amplificar utilizando los siguientes parámetros de ciclado:
| Etapa del ciclo | Temperatura | Velocidad de cambio | Duración | N.º de ciclos |
|---|---|---|---|---|
| Desnaturalización inicial | 94 °C | max | 3 min | 1 |
| Desnaturalización Descenso hacia la temperatura de hibridación Hibridación Extensión | 94 °C de 66 °C a 58 °C 58 °C 65 °C | max 0,2 °C/s max max | 30 s 40 s 50 s 6 min | 4 |
| Extensión final | 65 °C | max | 10 min | 1 |
| Mantenimiento | 4 °C | - | ∞ | - |
El siguiente esquema muestra las etapas y condiciones térmicas del ciclado. 
Transferir la muestra a un tubo nuevo Eppendorf DNA Lobind de 1,5 ml.
Resuspender las microesferas AMPure XP mediante vórtex.
Añadir 40 μl de microesferas resuspendidas AMPure XP a la reacción y mezclar dando suaves golpes al tubo con el dedo.
Incubar en el Hula mixer (o mezclador rotatorio) durante 5 min a temperatura ambiente.
Preparar 500 µl de etanol al 80 % con agua sin nucleasas.
Centrifugar las muestras y sedimentar las microesferas en un imán hasta que el eluido se vuelva claro e incoloro. Sin quitar el tubo del imán, retirar el sobrenadante con una pipeta.
Mantener el tubo en el imán, lavar las microesferas con 200 µl de etanol al 80 %, sin desplazar el sedimento. Retirar el etanol con una pipeta y desechar.
Repetir el paso anterior.
Centrifugar brevemente y colocar el tubo de nuevo en el imán. Retirar con una pipeta cualquier residuo de etanol. Dejar secar el sedimento durante 30 s aproximadamente, sin dejar que se agriete.
Quitar el tubo de la gradilla magnética y resuspender el sedimento en 10 µl de agua sin nucleasas. Centrifugar e incubar durante 2 min a temperatura ambiente.
Sedimentar las microesferas en un imán, durante al menos 1 minuto, hasta que el eluido se vuelva claro e incoloro.
Extraer y guardar 10 µl de eluido en un tubo nuevo Eppendorf DNA Lobind de 1,5 ml.
Transferir 10 µl de ADNc biotinilado al paso de captura.
4. Captura
Material
- 10 μl de ADNc biotinilado
Consumibles
- Tris-HCl 1 M, pH 7,5
- NaCl 5 M (Sigma, 71386)
- EDTA 0,5 M, pH 8 (Thermo Scientific, R1021)
- Streptavidina M280, 10 μg/μl (Invitrogen, 11205D)
- Nuclease-free water (e.g. Thermo Scientific, AM9937)
- Tubos Falcon de 15 ml
- 1.5 ml Eppendorf DNA LoBind tubes
- 0.2 ml thin-walled PCR tubes
Instrumental
- Vortex mixer
- Microfuge
- Hula mixer (gentle rotator mixer)
- Gradilla magnética (p. ej., Invitrogen DynaMag-2 Magnet, 12321D)
- Ice bucket with ice
- P1000 pipette and tips
- P200 pipette and tips
- P100 pipette and tips
- P20 pipette and tips
- P2 pipette and tips
Preparar 200 μl de tampón Tris-HCl 10 mM con pH 7,5 para utilizar más adelante.
Preparar 4 ml de tampón de lavado/ligación 2×, con la siguiente composición: 10 mM Tris-HCl pH 7,5; 2 M NaCl; 1 mM EDTA.
| Rectivo | Concentración inicial | Concentración final | Volumen |
|---|---|---|---|
| Tris-HCl pH 7,5 | 1 M | 10 mM | 40 μl |
| NaCl | 5 M | 2 M | 1 600 μl |
| EDTA | 0,5 M | 1 mM | 8 μl |
| Agua sin nucleasas | - | - | 2 352 μl |
| Total | - | - | 4 000 μl |
Transferir 3,5 ml del tampón de lavado/ligación 2× (10 mM Tris-HCl pH 7,5; 2 M NaCl; 1 mM EDTA) a un tubo Falcon nuevo de 15 ml.
Añadir 3,5 ml de agua sin nucleasas al mismo tubo Falcon de 15 ml, con lo que se obtendrá un volumen final de 7 ml de tampón de lavado/ligación 1×.
Resuspender las microesferas M280 de streptavidina (10 μg/μl) mediante agitación en vórtex.
Transferir 5 μl de microesferas de streptavidina a un tubo nuevo Eppendorf DNA Lobind de 1,5 ml.
Añadir 1 ml de tampón de lavado/ligación 1× y agitar en vórtex las microesferas con el tampón durante 5 s.
Centrifugar brevemente el tubo, sedimentar las microesferas en un imán durante 2 min y retirar el sobrenadante con la pipeta.
Repetir los pasos 7 y 8 dos veces más, hasta realizar 3 lavados en total.
Es fundamental utilizar tampón 2× en el paso siguiente. Utilizar tampón 1× dará lugar a una unión deficiente.
Resuspender las microesferas en 10 μl de tampón de lavado/ligación 2× hasta alcanzar una concentración final de 5 μg/μl.
Añadir 10 μl de microesferas preparadas a una concentración de 5 μg/μl (50 μg en total) en el tubo con 10 μl de ADNc biotinilado.
Incubar en el hula mixer (o mezclador rotatorio) durante 20 min a temperatura ambiente.
En los siguientes pasos, es importante sedimentar las microesferas en el imán durante los tiempos indicados para asegurarse de que no queden en la solución, ya que son difíciles de ver.
Añadir 1 ml de tampón de lavado/ligación 1× y agitar en vórtex el ADN y las microesferas con el tampón durante 5 s.
Centrifugar brevemente el tubo, sedimentar las microesferas en un imán durante 3 min y retirar el sobrenadante con la pipeta. Procure no aspirar las microesferas.
Repetir los pasos 13 y 14 dos veces más, hasta realizar 3 lavados en total.
Añadir 200 μl de 10 mM Tris-HCl, pH 7,5, y agitar las microesferas en vórtex durante 5 s.
Centrifugar brevemente y colocar el tubo de nuevo en la gradilla magnética durante 3 min. Retirar el sobrenadante con la pipeta.
Quitar el tubo de la gradilla y resuspender el sedimento en 20 µl de agua sin nucleasas.
Agitar en vórtex durante 5 s y centrifugar brevemente hasta recoger el conjugado de amplicón y microesferas.
Transferir 20 μl del conjugado al paso de PCR posterior a la captura.
5. PCR posterior a la captura
Material
- 20 μl de conjugado amplicón-microesferas
- Cebadores de PCR (PRM)
Consumibles
- LongAmp Hot Start Taq 2X Master Mix (NEB, M0533)
- Microesferas Agencourt AMPure XP (Beckman Coulter™, A63881)
- Kit Qubit 1x dsDNA HS (ThermoFisher, Q33230)
- Kit Agilent Technologies DNA 12000
- Etanol al 80 % recién preparado con agua sin nucleasas
- Agua sin nucleasas (p. ej., ThermoFisher, AM9937)
- 0.2 ml thin-walled PCR tubes
- 1.5 ml Eppendorf DNA LoBind tubes
- Qubit™ Assay Tubes (Invitrogen, Q32856)
Instrumental
- Thermal cycler
- Vortex mixer
- Hula mixer (rotator mixer)
- Gradilla magnética (p. ej., Invitrogen DynaMag-2 Magnet, 12321D)
- Microfuge
- Ice bucket with ice
- P1000 pipette and tips
- P200 pipette and tips
- P100 pipette and tips
- P20 pipette and tips
- P10 pipette and tips
- P2 pipette and tips
- Qubit™ fluorometer (or equivalent for QC check)
Equipo opcional
- Bioanalizador Agilent (o equivalente)
Descongelar los cebadores de PCR (PRM) a temperatura ambiente, centrifugar y colocar en hielo.
En un tubo de PCR de pared fina (0,2 ml), preparar la siguiente mezcla de reacción de PCR:
| Reactivo | Concentración inicial | Concentración final | Volumen |
|---|---|---|---|
| Cebadores de PCR (PRM) | 10 μM | 0,2 μM | 1 μl |
| Agua sin nucleasas | - | - | 4 μl |
| Mezcla maestra 2X LongAmp Hot Start Taq | 2× | 1× | 25 μl |
| Total | - | - | 30 μl |
Mezclar con la pipeta.
Resuspender el conjugado de amplicón y microesferas con la pipeta y transferir 20 μl al tubo de PCR que contiene la mezcla de reacción. Mezclar con la pipeta.
Evitar que el conjugado de amplicón y microesferas sedimente antes de cargarlo en el termociclador.
Conviene no dejar el conjugado en reposo durante mucho tiempo antes de cargarlo en el termociclador.
NO centrifugar el conjugado de amplicón y microesferas en ningún caso.
No centrifugar el tubo; transferir de inmediato al termociclador y amplificar mediante las siguientes etapas de ciclado:
| Etapas | Temperatura | Duración | Nº de ciclos |
|---|---|---|---|
| Desnaturalización inicial | 94 °C | 3 min | 1 |
| Desnaturalización Hibridación Extensión | 94 °C 56 °C 65 °C | 15 s 15 s 6 min | 4 |
| Extensión final | 65 °C | 10 min | 1 |
| Conservación | 4 °C | ∞ | - |
Resuspender las microesferas AMPure XP mediante vórtex.
Transferir la muestra a un tubo nuevo Eppendorf DNA Lobind de 1,5 ml.
Añadir 40 μl de microesferas resuspendidas AMPure XP a la mezcla de reacción y dar suaves golpes al tubo con el dedo.
Incubar en el hula mixer (o mezclador rotatorio) durante 5 min a temperatura ambiente.
Preparar 500 µl de etanol al 80 %, con agua sin nucleasas.
Centrifugar la muestra y sedimentar en una gradilla magnética hasta que el sobrenadante se vuelva claro e incoloro. Sin quitar el tubo de la gradilla, retirar el sobrenadante con una pipeta.
Mantener el tubo en el imán, lavar las microesferas con 200 µl de etanol al 80 % recién preparado, sin desplazar el sedimento. Retirar el etanol con una pipeta y desechar.
Repetir el paso anterior.
Centrifugar brevemente y colocar el tubo de nuevo en la gradilla magnética. Retirar con una pipeta cualquier residuo de etanol. Dejar secar el sedimento durante 30 s aproximadamente, sin dejar que se agriete.
Quitar el tubo de la gradilla magnética y resuspender el sedimento en 15 µl de agua sin nucleasas.
Sedimentar las microesferas en un imán hasta que el eluido se vuelva claro e incoloro.
Extraer y guardar 15 µl de eluido en un tubo nuevo Eppendorf DNA Lobind de 1,5 ml.
Deshechar las microesferas sedimentadas.
Cuantificar 1 μl de muestra eluida con un fluorímetro Qubit - se espera recuperar una cantidad superior a 100 ng.
Cuantificar 200 fmol de ADNc a partir del tamaño medio de fragmento, identificado con un Bioanalyzer de Agilent.
En su defecto, se asume un tamaño medio de fragmento de 1kbp.
Imagen: ejemplo de distribución de la longitud de fragmento de amplicones: 3 reproducciones biológicas de ADNc 3’ de PBMCs, preparado con el ensayo Chromium Next GEM Universal 3’ Gene Expression (V3.1) de 10x Genomics y procesadas con el protocolo de ADNc 3’ 10x de Oxford Nanopore Technologies. En este caso, se han analizado los amplicones con el Bioanalyzer 2100 y el kit DNA 12000.
Transferir 200 fmol de ADNc al paso de preparación de extremos.
6. Preparación de extremos
Material
- 200 fmol de amplicones de ADNc
- AMPure XP Beads (AXP)
Consumibles
- Mezcla enzimática NEBNext® Ultra II End Prep, del módulo NEBNext® Ultra II End Repair (NEB, E7546)
- Tampón de reacción NEBNext® Ultra II End Prep, del módulo NEBNext® Ultra II End Repair (NEB, E7546)
- Kit Qubit dsDNA HS (Invitrogen Q32851)
- Nuclease-free water (e.g. Thermo Scientific, AM9937)
- Etanol al 80 % recién preparado con agua sin nucleasas
- 0.2 ml thin-walled PCR tubes
- 1.5 ml Eppendorf DNA LoBind tubes
- Qubit™ Assay Tubes (Invitrogen, Q32856)
Instrumental
- P1000 pipette and tips
- P100 pipette and tips
- P10 pipette and tips
- Thermal cycler
- Microfuge
- Hula mixer (gentle rotator mixer)
- Gradilla magnética
- Ice bucket with ice
- Qubit™ fluorometer (or equivalent for QC check)
Preparar los reactivos NEBNext Ultra II End Repair/dA-tailing Module de acuerdo con las instrucciones del fabricante y poner en hielo:
Para obtener un rendimiento óptimo, NEB recomienda lo siguiente:
Descongelar todos los reactivos en hielo.
Mezclar bien cada uno de ellos.
Note: no mezclar en vórtex la mezcla Ultra II End Prep Enzyme Mix.Centrifugar los tubos antes de la primera apertura del día.
El tampón de reacción NEBNext Ultra II End Prep tal vez contenga un precipitado blanco. En tal caso, dejar que la mezcla alcance la temperatura ambiente y pipetear el tampón varias veces para dispersar el precipitado. Después, agitar brevemente en vórtex.
Transferir 200 fmol de amplicón de ADN a un tubo nuevo de PCR de 0,2 ml y ajustar el volumen a 50 µl con agua sin nucleasas.
En el mismo tubo de PCR, mezclar lo siguiente:
Entre cada adición, mezclar con la pipeta de 10 a 20 veces.
| Reactivo | Volumen |
|---|---|
| Amplicón ADNc | 50 µl |
| Ultra II End-prep Reaction Buffer | 7 µl |
| Ultra II End-prep Enzyme Mix | 3 µl |
| Total | 60 µl |
Mezclar bien la reacción con la pipeta y centrifugar brevemente.
Con ayuda de un termociclador, primero incubar a 20 °C durante 5 minutos, luego a 65 °C durante 5 minutos y después mantener a 4 °C.
Transferir la muestra de ADN a un tubo nuevo Eppendorf DNA Lobind de 1,5 ml.
Resuspender las microesferas AMPure XP (AXP) mediante vórtex.
Añadir 60 µl de microesferas AMPure XP (AXP) resuspendidas a la mezcla de preparación de extremos y mezclar golpeando suavemente el tubo con el dedo.
Incubar en el hula mixer (o mezclador rotatorio) durante 5 min a temperatura ambiente.
Preparar 500 µl de etanol al 80 % con agua sin nucleasas.
Centrifugar la muestra y sedimentar en una gradilla magnética hasta que el sobrenadante se vuelva claro e incoloro. Sin mover el tubo, retirar el sobrenadante con una pipeta.
Mantener el tubo en la gradilla, lavar las microesferas con 200 µl de etanol al 80 % sin desplazar el sedimento. Retirar el etanol con una pipeta y desechar.
Repetir el paso anterior.
Centrifugar y colocar el tubo de nuevo en la gradilla magnética. Retirar con una pipeta cualquier residuo de etanol. Dejar secar el sedimento durante 30 s aproximadamente, sin dejar que se agriete.
Quitar el tubo de la gradilla y resuspender el sedimento en 61 µl de agua sin nucleasas, mediante suaves movimientos de la pipeta o dando ligeros golpes en el tubo con el dedo. Incubar durante 2 min a temperatura ambiente.
Sedimentar las microesferas en la gradilla magnética, durante al menos 1 minuto, hasta que el eluido se vuelva claro e incoloro.
Extraer y guardar 61 µl de eluido en un tubo nuevo Eppendorf DNA Lobind de 1,5 ml.
Cuantificar 1 μl de muestra eluida con un fluorímetro Qubit.
Tomar el ADN reparado y proceder a la ligación de los adaptadores. En este momento también se puede guardar la muestra a 4 °C hasta el día siguiente.
7. Ligación de los adaptadores y purificación
Material
- Ligation Adapter (LA)
- Ligation Buffer (LNB)
- Short Fragment Buffer (SFB)
- AMPure XP Beads (AXP)
- Elution Buffer (EB)
Consumibles
- Kit Qubit dsDNA HS (Invitrogen Q32851)
- Ligasa de ADN Salt-T4® (NEB, M0467)
- 1.5 ml Eppendorf DNA LoBind tubes
- Qubit™ Assay Tubes (Invitrogen, Q32856)
Instrumental
- Gradilla magnética
- Hula mixer (rotator mixer)
- Microfuge
- Vortex mixer
- P1000 pipette and tips
- P100 pipette and tips
- P20 pipette and tips
- P10 pipette and tips
- Qubit™ fluorometer (or equivalent for QC check)
Recomendamos utilizar la ligasa de ADN Salt-T4®(NEB, M0467).
Se adquiere por separado o con el módulo NEBNext® Companion v2 de secuenciación por ligación de Oxford Nanopore Technologies® (E7672S o E7672L).
La ligasa de ADN Quick T4 (NEB, E6057) incluida en la versión anterior del módulo NEBNext® Companion Module de secuenciación por ligación de Oxford Nanopore Technologies® (NEB, E7180S or E7180L) también es compatible, pero el nuevo reactivo recomendado ofrece una ligación más eficiente.
Aunque las ligasas de otros fabricantes incluyan su propio tampón, la eficiencia del adaptador —Ligation Adapter (LA)— es mayor cuando se usa el tampón Ligation Buffer (LNB) incluido en el kit Ligation Sequencing.
Centrifugar brevemente el adaptador —Ligation Adapter (LA)— y la ligasa de ADN Salt-T4® y poner en hielo.
Descongelar el vial Ligation Buffer (LNB) a temperatura ambiente, centrifugar y mezclar con la pipeta. Debido a su viscosidad, la agitación en vórtex de este tampón es ineficaz. Tras descongelar y mezclar, colocar en hielo de inmediato.
Descongelar el vial Elution Buffer (EB) a temperatura ambiente y mezclar en vórtex. Centrifugar brevemente y poner en hielo.
Descongelar el reactivo Short Fragment Buffer (SFB) a temperatura ambiente y mezclar en vórtex. Centrifugar brevemente y poner en hielo.
En un tubo Eppendorf DNA LoBind de 1,5 ml, mezclar en el siguiente orden:
| Reactivo | Volumen |
|---|---|
| Muestra de ADN del paso anterior | 60 µl |
| Ligation Adapter (LA) | 5 µl |
| Ligation Buffer (LNB) | 25 µl |
| Ligasa de ADN Salt-T4® | 10 µl |
| Total | 100 µl |
Mezclar bien la reacción con la pipeta y centrifugar brevemente.
Incubar la reacción durante 10 min a temperatura ambiente.
Resuspender las microesferas AMPure XP Beads (AXP) mediante vórtex.
Añadir 40 μl de microesferas AMPure XP (AXP) resuspendidas a la mezcla de reacción y dar suaves golpes al tubo con el dedo.
Incubar en el hula mixer (o mezclador rotatorio) durante 5 min a temperatura ambiente.
Centrifugar brevemente la muestra y sedimentar en una gradilla magnética. Cuando el sobrenadante esté claro e incoloro, retirarlo con una pipeta.
Lavar las microesferas con 250 μl de Short Fragment Buffer (SFB). Golpear el tubo suavemente con el dedo, centrifugar brevemente, colocar de nuevo en la gradilla y dejar que las microesferas sedimenten. Retirar el sobrenadante con una pipeta y desechar.
Nota: retirar el sobrenadante con cuidado; la viscosidad del tampón tal vez contribuya a que se pierdan microesferas del sedimento.
Repetir el paso anterior.
Centrifugar y colocar el tubo de nuevo en la gradilla magnética. Retirar con una pipeta cualquier residuo de sobrenadante. Dejar secar el sedimento durante 30 s aproximadamente, sin dejar que se agriete.
Quitar el tubo de la gradilla magnética y resuspender el sedimento en 34 µl de Elution Buffer (EB).
Centrifugar brevemente la muestra e incubar durante 10 min a temperatura ambiente.
Sedimentar las microesferas en la gradilla magnética, durante al menos 1 minuto, hasta que el eluido se vuelva claro e incoloro.
Extraer y conservar 34 μl del eluido que contiene la biblioteca de ADN en un tubo nuevo Eppendorf DNA LoBind de 1,5 ml.
Cuantificar 1 μl de muestra eluida utilizando un fluorímetro Qubit.
Diluir entre 50 y 100 fmol de biblioteca preparada en 32 μl de Elution Buffer (EB).
En su defecto, asumir una longitud media de fragmento de 1 kbp y continuar con 33 ng de biblioteca.
Nota: si la biblioteca de ADN está por debajo de la concentración requerida, utilizar el volumen completo, 32 μl de ADN eluido, para secuenciar.
Atención tenga en cuenta que una recuperación muy baja en ocasiones se debe a un fallo en la preparación de la biblioteca.
Si es necesario, recomendamos utilizar una calculadora de masa a moles como la calculadora de NEB.
Se recomienda cargar entre 50 y 100 fmol de esta última biblioteca, en la celda de flujo R10.4.1.
Cargar la concentración recomendada garantizará una ocupación de poros óptima y un elevado rendimiento de secuenciación. Si es necesario, diluir la biblioteca en Elution Buffer (EB).
La biblioteca preparada se usará para cargar la celda de flujo. Conservarla en hielo o a 4 °C hasta el momento de cargar.
Recomendaciones de almacenamiento de la biblioteca
Recomendamos guardar las bibliotecas en tubos Eppendorf DNA LoBind a 4 °C, durante periodos de tiempo cortos o en caso de uso repetido, por ejemplo, para recargar celdas de flujo entre lavados. Para uso individual y almacenamiento a largo plazo por periodos de más de 3 meses, recomendamos guardar las bibliotecas a −80 °C en tubos Eppendorf DNA LoBind.
8. Acondicionamiento y carga de celdas de flujo PromethION
Material
- Sequencing Buffer (SB)
- Library Beads (LIB)
- Library Solution (LIS)
- Flow Cell Tether (FCT)
- Flow Cell Flush (FCF)
Consumibles
- Celdas de flujo PromethION
- 1.5 ml Eppendorf DNA LoBind tubes
Instrumental
- Dispositivo PromethION
- Pantalla protectora celdas de flujo PromethION
- P1000 pipette and tips
- P200 pipette and tips
- P20 pipette and tips
Este kit es compatible solo con celdas de flujo R10.4.1 (FLO-PRO114M).
Al sacar las celdas de flujo de la nevera, esperar 20 min antes de insertarlas en el PromethION, hasta que hayan alcanzado la temperatura ambiente. En ambientes húmedos, en ocasiones se forma condensación en la celda de flujo. Inspeccione las clavijas doradas del conector, situadas en la parte superior e inferior de la celda de flujo, en busca de condensación y si la hubiera, límpiela con una toallita sin pelusa. Compruebe que haya una almohadilla térmica negra enganchada en la parte posterior de la celda de flujo.
Descongelar los viales Sequencing Buffer (SB), Library Beads (LIB) o Library Solution (LIS) —si se requiere— Flow Cell Tether (FCT) y un tubo de Flow Cell Flush (FCF) a temperatura ambiente y mezclar en vórtex. Centrifugar brevemente y poner en hielo.
Preparar la mezcla de acondiciomiento, combinando Flow Cell Tether (FCT) y Flow Cell Flush (FCF) como se indica a continuación. Agitar en vórtex a temperatura ambiente.
En un tubo nuevo y adecuado al número de celdas de flujo que se vayan a utilizar, combinar los siguientes reactivos:
| Reactivo | Volumen por celda de flujo |
|---|---|
| Flow Cell Flush (FCF) | 1 170 µl |
| Flow Cell Tether (FCT) | 30 µl |
| Volumen total | 1 200 µl |
Cargar la(s) celda(s) de flujo en el/los puerto(s) de acople del PromethION 24/48 de la siguiente manera:
- Alinear la celda de flujo con el conector (horizontal y verticalmente) antes de insertarla suavemente en la posición.
- Presionar la celda de flujo hacia abajo con firmeza hasta que el cierre encaje y haga clic.


Insertar la celda de flujo en el ángulo equivocado puede dañar las clavijas del PromethION y afectar los resultados de secuenciación. Si observa que las clavijas de las posiciones del PromethION están dañadas, contacte con el servicio de asistencia en support@nanoporetech.com.

Realice una comprobación de la celda de flujo para evaluar el número de poros disponibles antes de cargar la biblioteca.
Este paso puede omitirse si la celda de flujo se ha comprobado con anterioridad. Encontrará más información en el documento de comprobación de la celda de flujo.
Deslizar la tapa del puerto de entrada en el sentido de las agujas del reloj.

Tenga cuidado al extraer tampón de la celda de flujo. No retire más de 20-30 μl y compruebe que el tampón cubra la matriz de poros en todo momento. La introducción de burbujas de aire en la matriz puede dañar los poros de manera irreversible.
Tras abrir el puerto de entrada, retirar una pequeña cantidad de tampón para quitar las burbujas de aire:
- Ajustar una pipeta P1000 a 200 μl.
- Introducir la punta de la pipeta en el puerto de entrada.
- Girar la rueda hasta que el indicador de volumen marque 220-230 μl o hasta que se pueda ver una pequeña cantidad de tampón entrar en la punta de la pipeta.

Cargar 500 μl de mezcla de acondicionamiento por el puerto de entrada, evitando introducir burbujas de aire. Esperar 5 min. Durante este tiempo, preparar la biblioteca para cargar siguiendo los siguientes pasos del protocolo.

Mezclar minuciosamente con la pipeta el contenido del vial Library Beads (LIB).
El vial Library Beads (LIB) contiene microesferas en suspensión. Las microesferas sedimentan muy rápido; por eso es crucial mezclarlas justo antes de su uso.
Recomendamos utilizar Library Beads (LIB) en la mayoría de experimentos de secuenciación. No obstante, el reactivo Library Solution (LIS) está disponible en caso de utilizar bibliotecas más viscosas.
En un tubo Eppendorf DNA LoBind de 1,5 ml preparar la biblioteca de la siguiente manera:
| Reactivo | Volumen por celda de flujo |
|---|---|
| Sequencing Buffer (SB) | 100 µl |
| Library Beads (LIB) bien mezclado antes de su uso | 68 µl |
| Biblioteca de ADN | 32 µl |
| Total | 200 µl |
Nota: Hemos aumentado el volumen de carga de la biblioteca para mejorar la cobertura de la matriz.
Cargar 500 μl de mezcla de acondicionamiento por el puerto de entrada y así finalizar el acondicionamiento de la celda de flujo.

Mezclar la biblioteca suavemente con la pipeta antes de cargarla.
Con una pipeta p1000, cargar 200 μl de biblioteca por el puerto de entrada.

Cerrar la válvula y sellar con ello el puerto de entrada.

Colocar la pantalla protectora sobre la celda de flujo en cuanto se haya cargado la biblioteca, favorecerá un rendimiento de secuenciación óptimo.
Recomendamos dejar la pantalla protectora colocada mientras la celda de flujo esté cargada, incluyendo durante los lavados o recargas. La pantalla puede quitarse una vez se haya extraído la biblioteca de la celda de flujo.
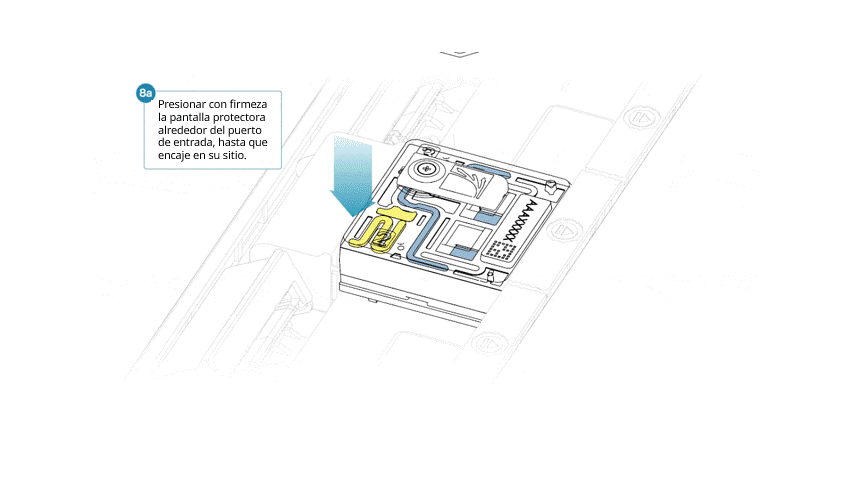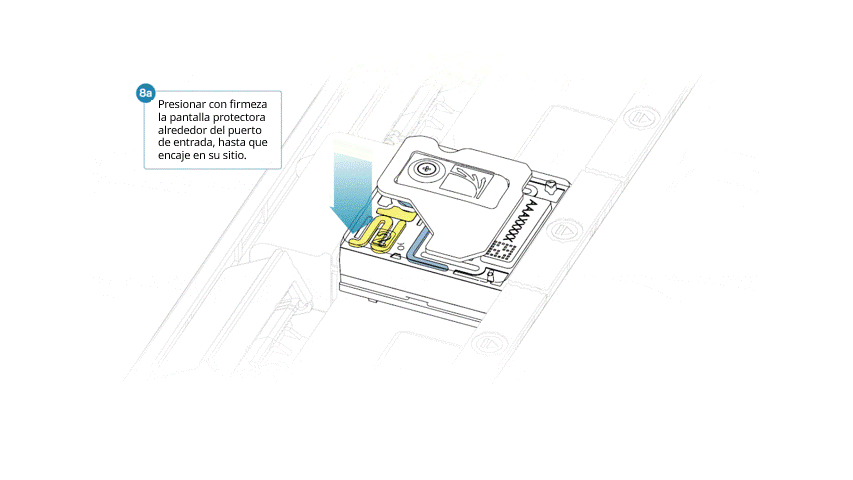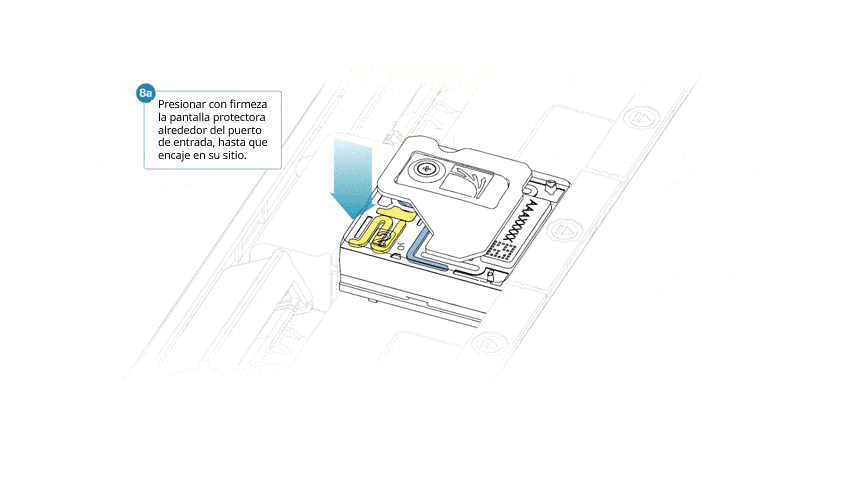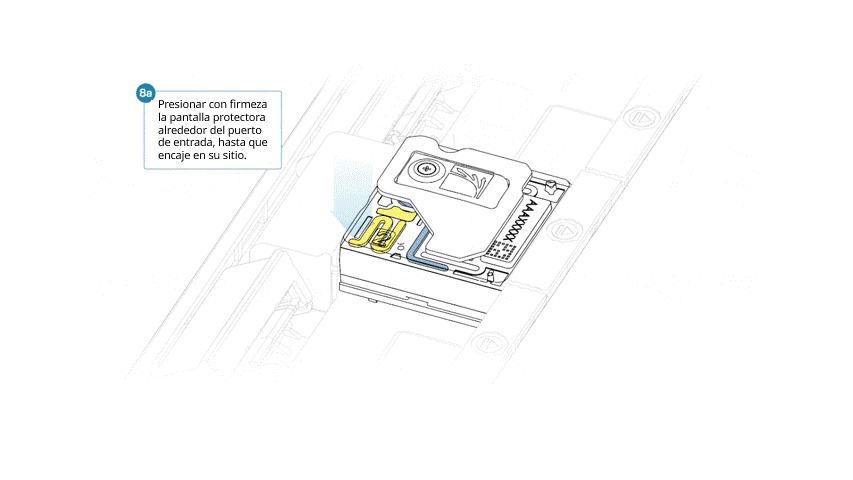
Si se ha retirado la pantalla protectora, colocar de nuevo de la siguiente manera:
- Alinear la abertura de la pantalla protectora con la tapa del puerto de entrada. El borde delantero debe quedar por encima del identificador de la celda de flujo.
- Presionar con firmeza la pantalla protectora alrededor de la tapa del puerto de entrada. Cuando el clip del puerto de entrada encaje bajo la tapa del mismo, se producirá un clic.

Cerrar la tapa del PromethION cuando esté listo para empezar un experimento de secuenciación en MinKNOW.
Esperar al menos 10 min tras cargar las celdas de flujo antes de iniciar cualquier experimento ayudará a aumentar el rendimiento de la secuenciación.
9. Adquisición de datos e identificación de bases
No recomendamos secuenciar y analizar datos simultáneamente en el mismo dispositivo.
A fin de que la capacidad de procesamiento mantenga el ritmo de exigencia de la secuenciación o el análisis, desaconsejamos ejecutar ambos procesos al mismo tiempo.
Compruebe que todo experimento de secuenciación haya finalizado antes de empezar a analizar datos. El análisis de datos se llevará a cabo tras la secuenciación.
Del mismo modo, tampoco recomendamos empezar un experimento de secuenciación mientras el dispositivo esté analizando datos.
Use siempre la versión más reciente del MinKNOW.
Recomendamos actualizar MinKNOW antes de empezar un experimento.
Dispone de instrucciones de actualización en el protocolo de MinKNOW.]
Cómo empezar a secuenciar
El programa MinKNOW gestiona el dispositivo de secuenciación y la adquisición de datos. Compruebe que MinKNOW esté instalado en su ordenador o dispositivo. Encontrará instrucciones adicionales para configurar experimentos de secuenciación en el Protocolo de MinKNOW.
Recomendamos configurar el experimento de secuenciación con las recomendaciones indicadas a continuación. Los demás parámetros se dejan en su configuración predeterminada.
Configuración de MinKNOW
Estas son las recomendaciones de configuración de MinKNOW:
Posiciones
Posición de celda de flujo: [definido por el usuario]
Nombre del experimento: [definido por el usuario]
Tipo de celda de flujo: FLO-PRO114M
ID de muestra: [definido por el usuario]
Kit
Selección del kit: Ligation Sequencing Kit (SQK-LSK114)
Configuración del experimento
Secuenciación y análisis
Identificación de bases: Activado
Bases modificadas: Desactivado [predeterminado]
Modelo: Identificación de bases de gran precisión (HAC)
Adición de códigos de barras: Desactivado [predeterminado]
Alineación: Desactivado [predeterminado]
Muestreo adaptativo: Desactivado [predeterminado]
Opciones avanzadas
Intervalo entre exploraciones de poros: 1,5 [predeterminado]
Reservar poros: Activado [predeterminado]
Ubicaciones de destino de los datos
Duración máxima de la ejecución: detener el experimento cuando la secuenciación alcance 72 h
Salida
Formatos de salida
.BAM: Desactivado
.FASTQ: Desactivado
Lecturas sin procesar: Activado [predeterminado]
.POD5: Activado [predeterminado]
Filtrado: Activado [predeterminado]
Índice Q score: 8 [predeterminado]
Longitud mínima de lectura: 200 b
Análisis de datos
Una vez la secuenciación haya terminado, es posible reutilizar o devolver la celda de flujo, tal como se describe en la sección Reutilización y retorno de celdas de flujo.
Tras secuenciar e identificar los nucleótidos, es posible analizar los datos, tal como se describe en la sección Análisis de datos.
10. Reutilización y retorno de celdas de flujo
Material
- Kit Flow Cell Wash (EXP-WSH004)
Una vez el experimento de secuenciación haya terminado, si quiere reutilizar la celda de flujo, siga las instrucciones del protocolo Flow Cell Wash Kit y guarde la celda de flujo a entre 2 °C y 8 °C.
El protocolo Flow Cell Wash Kit está disponible en la comunidad Nanopore.
Otra opción es seguir el procedimiento de devolución y enviarla de vuelta a Oxford Nanopore.
Aquí encontrará las instrucciones para devolver celdas de flujo.
Si tiene dudas o preguntas acerca del experimento de secuenciación, consulte la sección Resolución de problemas de la versión en línea de este protocolo.
11. Análisis de datos
Análisis de datos de la secuenciación transcriptómica de células individuales a partir de ADNc 3’
Los archivos de salida (FASTQ o BAM) se analizan con wf-single-cell, un flujo de trabajo bioinformático desarrollado en Nextflow que forma parte de la plataforma de análisis de datos EPI2ME de Oxford Nanopore Technologies. wf-single-cell está diseñado para ejecutarse desde la línea de comandos y desde EPI2ME Desktop Application. El flujo de trabajo wf-single-cell está disponible en GitHub.
Cómo ejecutar wf-single-cell
Para ejecutar el flujo de trabajo desde la línea de comandos:
nextflow run epi2me-labs/wf-single-cell \
--expected_cells <CELL NUMBER> \
--fastq <READS> \
--kit <KIT> \
--ref_genome_dir <REFERENCE> \
-profile standard
Donde expected _cells es el número de células seleccionadas en el experimento con 10x Chromium, fastq (o bam) es la ubicación de las lecturas identificadas, kit corresponde al kit y la versión de 10x utilizados (p. ej. 3prime:v4) y ref_genome_dir es la ubicación del genoma de referencia y los archivos de anotación de acuerdo con el formato de referencia 10x (encontrará más información en el centro de descargas de 10x.
Para ejecutar el flujo de trabajo, es necesario tener instalado Nextflow en el sistema y además, Docker o Singularity. Encontrará información adicional sobre cómo ejecutar los flujos de trabajo de EPI2ME desde la línea de comandos en la Guía de inicio rápido.
En la página de GitHub y en la documentación del flujo de trabajo se encuentra la información más reciente, junto con datos adicionales sobre cómo ejecutarlo y sobre los parámetros disponibles.
Archivos de salida
wf-single-cell genera un informe interactivo en formato HTML, que incluye parámetros básicos de control de calidad (puntuación de secuenciación, longitudes de lectura), un resumen del experimento (número de células, media de lecturas por célula, etc.), gráficos diagnósticos de saturación y de rodilla, así como gráficos UMAP para visualizar las agrupaciones celulares. También genera matrices de expresión génica y de transcritos en un formato compatible con las herramientas de análisis más utilizadas.
12. Problemas durante la extracción de ADN y preparación de la biblioteca
A continuación hay una lista de los problemas más frecuentes, con algunas posibles causas y soluciones propuestas.
También disponemos de una sección de preguntas frecuentes, FAQ, en la sección Support de la comunidad Nanopore.
Si ha probado las soluciones propuestas y los problemas aún persisten, póngase en contacto con el departamento de Asistencia técnica, bien por correo electrónico (support@nanoporetech.com) o a través del chat Live de la comunidad Nanopore.
Baja calidad de la muestra
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Baja pureza del ADN (la lectura del Nanodrop para ADN OD 260/280 es <1,8 y OD 260/230 es <2,0-2,2) | El método de extracción de ADN no proporciona la pureza necesaria | Los efectos de los contaminantes se muestran en el documento Contaminants document. Probar con otro método de extracción que no provoque el arrastre de contaminantes. Considere realizar un paso adicional de limpieza SPRI. |
Escasa recuperación de ADN tras la limpieza con microesferas AMPure
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Escasa recuperación | Pérdida de ADN debido a una proporción de microesferas AMPure por muestra inferior a lo previsto. | 1. Las microesferas AMPure precipitan con rapidez; antes de añadirlas a la muestra deben estar bien resuspendidas. 2. Si la proporción de microesferas AMPure por muestra es inferior a 0.4:1, los fragmentos de ADN, sean del tamaño que sean, se perderán durante la limpieza. |
| Escasa recuperación | Los fragmentos de ADN son más cortos de lo esperado | Cuanto menor sea la proporción de microesferas AMPure por muestra, más rigurosa será la selección de fragmentos largos frente a los cortos. Determinar siempre la longitud de la muestra de ADN en un gel de agarosa u otros métodos de electroforesis en gel y a continuación, calcular la cantidad adecuada de microesferas AMPure que se debe utilizar. 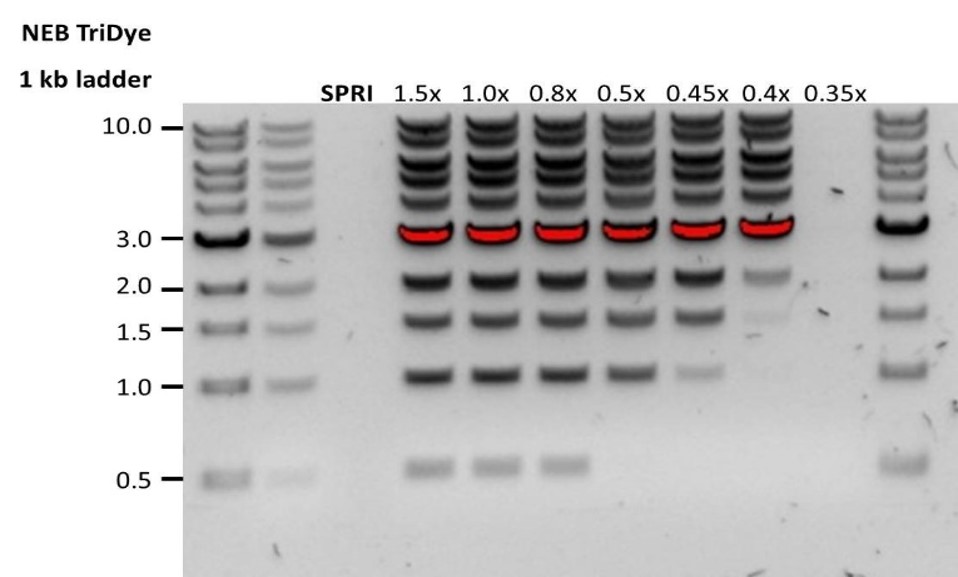 |
| Escasa recuperación tras la preparación de extremos | El paso de lavado utilizó etanol en una concentración inferior al 70 % | Si se utiliza etanol a una concentración inferior al 70 %, el ADN se eluye de las microesferas. Utilice el porcentaje correcto. |
13. Problemas durante el experimento de secuenciación
A continuación hay una lista de los problemas más frecuentes, con algunas posibles causas y soluciones propuestas.
También disponemos de una sección de preguntas frecuentes, FAQ, en la sección Support de la comunidad Nanopore.
Si ha probado las soluciones propuestas y los problemas aún persisten, póngase en contacto con el departamento de Asistencia técnica, bien por correo electrónico (support@nanoporetech.com) o a través del chat Live de la comunidad Nanopore.
Menos poros al inicio de la secuenciación que tras la evaluación de la celda de flujo
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| MinKNOW presentó al inicio de la secuenciación un número de poros inferior al indicado durante la evaluación de la celda de flujo. | Se introdujo una burbuja de aire en la matriz de nanoporos. | Tras comprobar el número de poros presente en la celda de flujo y antes de acondicionarla, es imprescindible quitar las burbujas de aire que haya cerca del puerto de purgado. Si no se quitan, las burbujas de aire pueden desplazarse a la matriz de nanoporos y dañar de manera irreversible los nanoporos expuestos al aire. En este vídeo se muestran algunas buenas prácticas para evitar que esto ocurra. |
| MinKNOW presentó al inicio de la secuenciación un número de poros inferior al indicado durante la evaluación de la celda de flujo. | La celda de flujo no está colocada correctamente. | Detener el ciclo de secuenciación, quitar la celda de flujo del dispositivo e insertarla de nuevo. Comprobar que está firmemente asentada en el dispositivo y que ha alcanzado la temperatura deseada. Si procede, probar con una posición diferente del dispositivo (GriION/PromethION). |
| MinKNOW presentó al inicio de la secuenciación un número de poros inferior al indicado durante la evaluación de la celda de flujo | La presencia de contaminantes en la biblioteca ha dañado o bloqueado los poros. | El recuento de poros durante la evaluación de la celda de flujo se efectúa utilizando las moléculas de ADN del control de calidad presentes en el tampón de almacenamiento. Al inicio de la secuenciación, se utiliza la misma biblioteca para calcular el número de poros activos. Por este motivo, se estima que puede haber una variabilidad del 10 % en el número de poros detectados. Tener un número de poros considerablemente inferior al inicio de la secuenciación puede deberse a la presencia de contaminantes en la biblioteca que hayan dañado las membranas o bloqueado los poros. Para mejorar la pureza del material de entrada tal vez sea necesario usar métodos de purificación o extracción de ADN/ARN alternativos. Los efectos de los contaminantes se muestran en la página Contaminants. Probar con un método de extracción diferente que no provoque el arrastre de contaminantes. |
Error en el script de MinKNOW
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| MinKNOW muestra el mensaje "Error en el script" | Reiniciar el ordenador y reiniciar MinKNOW. Si el problema persiste, reúna los archivos de registro de MinKNOW y contacte con el departamento de Asistencia técnica. Si no dispone de otro dispositivo de secuenciación, recomendamos que guarde la celda de flujo cargada a 4 °C y contacte con el servicio de asistencia técnica, que proporcionará instrucciones de almacenamiento adicionales. |
Ocupación de poro por debajo del 40 %
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Ocupación de poro inferior al 40 % | No se cargó suficiente cantidad de biblioteca en la celda de flujo | Cargue la biblioteca al volumen y la concentración adecuados, tal como se indica en el protocolo correspondiente. Cuantificar la biblioteca antes de cargarla, calcular moles con herramientas como la calculadora Biomath de Promega, y escoger la opción "dsDNA: μg to pmol". |
| Ocupación de poro próxima a 0 | Se utilizó el kit de secuenciación por ligación y los adaptadores de secuenciación no se ligaron al ADN | En la fase de ligación del adaptador, añadir la cantidad recomendada de NEBNext Quick Ligation Module (E6056) y el tampón Ligation Buffer (LNB) suministrado en el kit de secuenciación. Preparar una biblioteca de control con lambda para valorar la integridad de los reactivos de otros fabricantes. |
| Ocupación de poro próxima a 0 | Se utilizó el kit Ligation Sequencing y en la fase de lavado, después de la ligación del adaptador, se utilizó etanol en lugar de Long Fragment Buffer (LFB) o Short Fragment Buffer (SFB). | El etanol puede desnaturalizar la proteína motor en los adaptadores de secuenciación. Usar Long Fragment Buffer (LFB) o Short Fragment Buffer (SFB) después de la ligación de los adaptadores. |
| Ocupación de poro próxima a 0 | No hay anclaje en la celda de flujo | Los anclajes se añaden durante el acondicionamiento de la celda de flujo (viales (FLT)/(FCT)). Añadir Flush Tether (FLT) o Flow Cell Tether (FCT) al vial Flush Buffer (FB) o Flow cell Flush (FCF) antes de acondicionar la celda de flujo. |
Longitud de lectura más corta de lo esperado
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Longitud de lectura más corta de lo esperado | Fragmentación no deseada de la muestra de ADN | La longitud de lectura refleja la longitud del fragmento de la muestra de ADN. La muestra de ADN se puede fragmentar durante la extracción y la preparación de la biblioteca. 1. Consulte la sección de buenas prácticas de extracción en la página Extraction Methods de la comunidad Nanopore. 2. Visualizar la distribución de la longitud de los fragmentos de las muestras de ADN en un gel de agarosa antes de proceder a la preparación de la biblioteca. 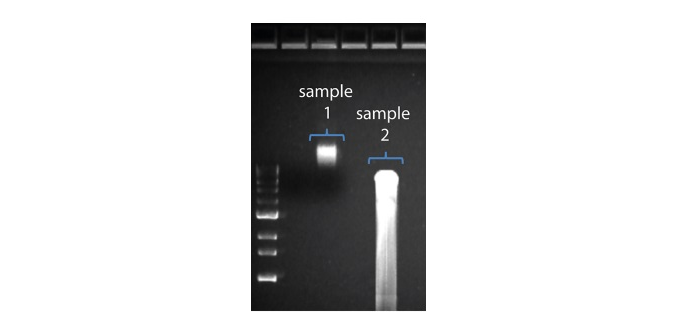 En la imagen superior, la muestra 1 contiene un elevado peso molecular, mientras que la muestra 2 se ha fragmentado. En la imagen superior, la muestra 1 contiene un elevado peso molecular, mientras que la muestra 2 se ha fragmentado.3. Durante la preparación de la biblioteca, evite pipetear y agitar en vórtex al mezclar los reactivos. Dar suaves golpes con el dedo o invertir el vial es suficiente. |
Gran proporción de poros no disponibles
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
Gran proporción de poros no disponibles (se muestran en azul en el panel de canales y en el gráfico de actividad de poros) 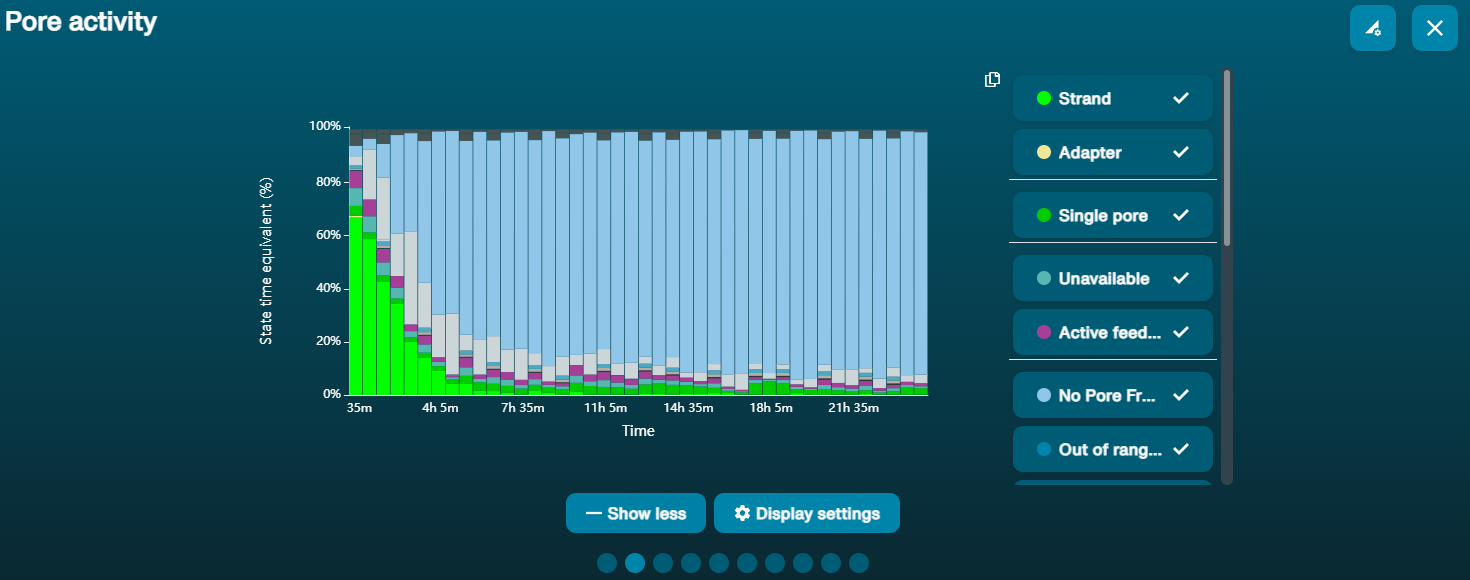 El gráfico de actividad de poros de arriba muestra una proporción creciente de poros "no disponibles» conforme pasa el tiempo. El gráfico de actividad de poros de arriba muestra una proporción creciente de poros "no disponibles» conforme pasa el tiempo. | Hay contaminantes presentes en la muestra | Algunos contaminantes se pueden eliminar de los poros mediante la función de desbloqueo incorporada en MinKNOW. Si funciona, el estado de los poros cambiará a "sequencing pores". Si la porción poros no disponibles se mantiene elevada o aumenta: 1. Es posible realizar un lavado con nucleasa utilizando el kit Flow Cell Wash (EXP-WSH004), o 2. Realizar varios ciclos de PCR para intentar diluir cualquier contaminante que pueda estar causando problemas. |
Gran proporción de poros inactivos
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Gran proporción de poros inactivos o no disponibles (se muestran en azul claro en el panel de canales y en el gráfico de actividad de poros). Los poros o membranas están dañados de manera irreversible) | Se han introducido burbujas de aire en la celda de flujo | Las burbujas de aire introducidas durante el acondicionamiento de la celda y carga de la biblioteca podrían dañar los poros de forma permanente. La forma más eficaz de evitar que esto ocurra se muestra en el vídeo Cómo cargar celdas de flujo PromethION. |
| Gran proporción de poros inactivos o no disponibles | Hay contaminantes presentes en la muestra | Los efectos de los contaminantes se muestran en el documento Contaminants. Probar con un método de extracción alternativo que no provoque el arrastre de contaminantes. |
Fluctuación de la temperatura
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Fluctuación de la temperatura | La celda de flujo ha perdido contacto con el dispositivo | Comprobar que una almohadilla térmica cubra la placa metálica de la parte posterior de la celda de flujo. Reinsertar la celda de flujo y presionar hasta que las clavijas del conector estén firmemente en contacto con el dispositivo. Si el problema persiste, contacte con el servicio de Asistencia técnica. |
Error al intentar alcanzar la temperatura deseada
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| MinKNOW muestra el mensaje «Error al intentar alcanzar la temperatura deseada» | El dispositivo se colocó en una ubicación con una temperatura ambiente inferior a la media o con escasa ventilación (lo que provoca el sobrecalientamiento de las celdas de flujo). | MinKNOW establece un tiempo para que la celda de flujo alcance la temperatura fijada. Si se supera el tiempo establecido, aparecerá un mensaje de error, pero el experimento de secuenciación continuará. Secuenciar a una temperatura incorrecta puede llevar a una disminución en el rendimiento y generar índices de calidad Q score menores. Corregir la ubicación del dispositivo, procurando que esté a temperatura ambiente y con buena ventilación; a continuación, reiniciar el proceso en MinKNOW. |







