Rapid Sequencing Kit V14 - gDNA (SQK-RAD114) (RSE_9177_v114_revQ_12Dec2025)
MinION: Protocol
Rapid Sequencing Kit V14 - gDNA (SQK-RAD114) V RSE_9177_v114_revQ_12Dec2025
Este es el protocolo más sencillo y rápido para secuenciar ADN genómico.
Este protocolo:
- Tiene un tiempo de preparación de la biblioteca de aproximadamente 10 mins
- Incluye fragmentación
- No es necesario utilizar ligasa de otros fabricantes
- Prescinde de PCR
De uso exclusivo en investigación
FOR RESEARCH USE ONLY
Contents
Introducción al protocolo
Preparación de la biblioteca
Secuenciación y análisis de datos
- 5. Adquisición de datos e identificación de bases
- 6. Reutilización y devolución de celdas de flujo
- 7. Análisis de datos
Resolución de problemas
Descripción general
Este es el protocolo más sencillo y rápido para secuenciar ADN genómico.
Este protocolo:
- Tiene un tiempo de preparación de la biblioteca de aproximadamente 10 mins
- Incluye fragmentación
- No es necesario utilizar ligasa de otros fabricantes
- Prescinde de PCR
De uso exclusivo en investigación
1. Aspectos generales
Este es un producto de acceso anticipado
En este artículo encontrará más información sobre programas de acceso anticipado y fases de lanzamiento de productos.
Utilice siempre la versión más reciente del protocolo.
Introducción al Rapid Sequencing Kit V14 (SQK-RAD114)
Este protocolo contiene instrucciones para completar paso a paso la secuenciación rápida de ADN genómico usando el kit Rapid Sequencing Kit V14 (SQK-RAD114). Este kit usa la novísima química del kit 14, que está optimizada para preparar la biblioteca rápidamente, con un material de laboratorio mínimo.
Antes de empezar, se recomienda hacer un experimento de control con lambda para familiarizarse con la tecnología.
Pasos en el proceso de secuenciación:
Preparación del experimento
Pasos:
- Extraer el ADN, evaluar su longitud, cantidad y pureza. Para ello, dispone del protocolo Input DNA/RNA QC.
Los controles de calidad realizados durante el protocolo son fundamentales para garantizar el éxito del experimento. - Contar con el kit de secuenciación, el instrumental adecuado y los reactivos de otros fabricantes
- Si no se ha instalado todavía, descargar el programa MinKNOW, necesario para obtener y analizar los datos
- Comprobar la(s) celda(s) de flujo para asegurarse de que tiene(n) poros suficientes para realizar una buena secuenciación.
Preparación de la biblioteca
Pasos:
| Pasos prep. biblioteca | Proceso | Duración | Parada opcional |
|---|---|---|---|
| Tagmentación | Tagmentar el ADN usando el reactivo Fragmentation Mix | 5 minutos | - |
| Ligación de los adaptadores rápidos | Ligar los adaptadores de secuenciación a los extremos de ADN | 5 minutos | Se recomienda secuenciar la biblioteca tan pronto como se liguen los adaptadores |
| Preparación y carga de la celda de flujo | Acondicionar la celda de flujo y cargar la biblioteca de ADN en ella | 10 minutos | - |
Secuenciación y análisis
Pasos:
- Empezar el proceso de secuenciación usando el programa MinKNOW, que obtendrá datos crudos del dispositivo y los convertirá en lecturas de bases identificadas.
- Opcional: Iniciar el programa EPI2ME y seleccionar un proceso de trabajo para realizar análisis complementarios, p. ej., análisis metagenómicos o cartografía de farmacorresistencia
Compatibilidad del protocolo
Este protocolo debería usarse solo en combinación con los siguientes productos:
- Rapid Sequencing Kit V14 (SQK-RAD114)
- Control Expansion (EXP-CTL001) (material complementario de control)
- R10.4.1 MinION flow cells (FLO-MIN114) (celdas de flujo MinION R10.4.1)
- Flow Cell Wash Kit (EXP-WSH004) (kit de lavado de celdas de flujo)
- MinION Mk1D - Requisitos informáticos MinION Mk1D
- GridION - Requisitos informáticos GridION
2. Material y consumibles
Material
- 100-150 ng de ADN genómico de alto peso molecular
- Rapid Sequencing Kit V14 (SQK-RAD114)
Consumibles
- MinION/GridION Flow Cell
- Seroalbúmina bovina (BSA) (50 mg/ml) (p. ej., Invitrogen™ UltraPure™ BSA 50 mg/ml, AM2616)
- Nuclease-free water (e.g. Thermo Scientific, AM9937)
- 1.5 ml Eppendorf DNA LoBind tubes
- 0.2 ml thin-walled PCR tubes
Instrumental
- MinION or GridION device
- MinION/GridION Flow Cell Light Shield
- Microfuge
- P1000 pipette and tips
- P100 pipette and tips
- P20 pipette and tips
- P10 pipette and tips
- P2 pipette and tips
- Timer
- Termociclador o termobloque
Equipo opcional
- Qubit™ fluorometer (or equivalent for QC check)
Para este protocolo, necesitará ~100 ng de ADN genómico de alto peso molecular.
Se pueden usar cantidades de muestra menores pero el resultado de la secuenciación será reducido.
Cantidad de muestra inicial de ADN
Cómo realizar un control de calidad del ADN de la muestra inicial
Es importante que la muestra de ADN cumpla con los requisitos de cantidad y calidad. Usar demasiado ADN, poco o de mala calidad (p. ej., que esté muy fragmentado, que contenga ARN o contaminantes químicos), puede afectar a la preparación de la biblioteca.
Para realizar un control de calidad en la muestra de ADN, consulte el protocolo Input DNA/ RNA QC
Contaminantes químicos
Dependiendo de cómo se extraiga el ADN de la muestra cruda, ciertos contaminantes químicos pueden permanecer en el ADN purificado, lo cual afecta la eficacia de la preparación de la biblioteca y la calidad de la secuenciación. Encontrará más información sobre contaminantes en la página Contaminants de la comunidad Nanopore.
Reactivos de otros fabricantes
Oxford Nanopore Technologies ha probado y recomienda el uso de todos los reactivos de otros fabricantes citados en este protocolo. No se han evaluado otras alternativas.
Recomendamos preparar estos reactivos siguiendo las instrucciones del fabricante.
Verificar la celda de flujo
Antes de empezar el experimento de secuenciación, recomendamos verificar el número de poros disponibles, presentes en la celda de flujo. La comprobación deberá realizarse en las primeras 12 semanas desde su adquisición, si se trata de celdas de flujo MinION, GridION o PromethION, y en las primeras cuatro semanas tras la compra de celdas de flujo Flongle. Oxford Nanopore Technologies sustituirá cualquier celda de flujo con un número de poros inferior al indicado en la tabla siguiente, siempre y cuando el resultado se notifique dentro de los dos días siguientes a la comprobación y se hayan seguido las instrucciones de almacenamiento. Para verificar la celda de flujo, siga las instrucciones del documento Flow Cell Check.
| Celda de flujo | Número mínimo de poros activos cubierto por la garantía |
|---|---|
| Flongle | 50 |
| MinION/GridION | 800 |
| PromethION | 5000 |
Contenido del Rapid Sequencing Kit V14 (SQK-RAD114)
3. Preparación de la biblioteca
Material
- 100-150 ng de ADN genómico de alto peso molecular
- Rapid Adapter (RA)
- Adapter Buffer (ADB)
- Fragmentation Mix (FRA)
Consumibles
- Nuclease-free water (e.g. Thermo Scientific, AM9937)
- 1.5 ml Eppendorf DNA LoBind tubes
- 0.2 ml thin-walled PCR tubes
Instrumental
- Termociclador o termobloque
- P2 pipette and tips
- P10 pipette and tips
Verificar la celda de flujo
Antes de empezar a preparar la biblioteca, recomendamos verificar la celda de flujo para comprobar que tiene poros suficientes para realizar un buen experimento.
Encontrará más información en las instrucciones de comprobación de la celda de flujo.
Descongelar los componentes del kit a temperatura ambiente, centrifugar brevemente y mezclar con la pipeta, como se indica en la tabla a continuación:
| Reactivo | 1. Descongelar a temperatura ambiente | 2. Centrifugar brevemente | 3. Mezclar con la pipeta |
|---|---|---|---|
| Fragmentation Mix (FRA) | Descongelado | ✓ | ✓ |
| Rapid Adapter (RA) | Descongelado | ✓ | ✓ |
| Adapter Buffer (ADB) | Descongelado | ✓ | ✓ |
Una vez descongelado, colocar todos los viales del kit en hielo.
Preparar el ADN en agua sin nucleasas.
- Transferir 100-150 ng de ADN genómico a un tubo de 1,5 ml Eppendorf DNA LoBind
- Ajustar el volumen a un total de 10 µl con agua sin nucleasas
- Mezclar golpeando suavemente el tubo con el dedo para evitar cizalladuras indeseadas
- Centrifugar brevemente
En un tubo de PCR de pared fina (0,2 ml), mezclar lo siguiente:
| Reactivo | Volumen |
|---|---|
| 100-150 ng de plantilla de ADN | 10 μl |
| Fragmentation Mix (FRA) | 1 μl |
| Total | 11 μl |
Mezclar golpeando suavemente el tubo con el dedo y centrifugar brevemente.
Incubar el tubo a 30 ⁰C durante 2 minutos y a continuación a 80 ⁰C durante 2 minutos más. Colocar el tubo en hielo unos instantes para enfriarlo.
Una vez tagmentado el ADN, proceder a la ligación del adaptador.
En un tubo de 1,5 ml Eppendorf DNA LoBind, diluir el adaptador, Rapid Adapter (RA), como se indica a continuación y mezclar con la pipeta:
| Reactivo | Volumen |
|---|---|
| Rapid Adapter (RA) | 1,5 μl |
| Adapter Buffer (ADB) | 3,5 μl |
| Total | 5 μl |
Añadir 1 μl de Rapid Adapter (RA) diluido al ADN tagmentado.
Mezclar golpeando suavemente el tubo con el dedo y centrifugar brevemente.
Incubar la reacción durante 5 minutos a temperatura ambiente.
La biblioteca de ADN preparada se usará para cargar la celda de flujo. Conservar la biblioteca en hielo hasta el momento de cargar.
4. Acondicionar y cargar la celda de flujo MinION/GridION
Material
- Flow Cell Flush (FCF)
- Flow Cell Tether (FCT)
- Library Solution (LIS)
- Library Beads (LIB)
- Sequencing Buffer (SB)
Consumibles
- MinION/GridION Flow Cell
- Seroalbúmina bovina (BSA) (50 mg/ml) (p. ej., Invitrogen™ UltraPure™ BSA 50 mg/ml, AM2616)
- Nuclease-free water (e.g. Thermo Scientific, AM9937)
- 1.5 ml Eppendorf DNA LoBind tubes
Instrumental
- MinION or GridION device
- MinION/GridION Flow Cell Light Shield
- P1000 pipette and tips
- P100 pipette and tips
- P20 pipette and tips
- P10 pipette and tips
Nótese, este kit es compatible solo con las celdas de flujo R10.4.1 (FLO-MIN114).
Sacar la celda de flujo de la nevera y dejarla a temperatura ambiente durante 20 minutos, mejorará la visibilidad de la matriz durante el acondicionamiento y carga de la muestra.
Acondicionar y cargar la celda de flujo
Recomendamos mirar el vídeo Cómo cargar celdas de flujo MinION antes de realizar el primer experimento.
Descongelar los viales Sequencing Buffer (SB), Library Beads (LIB) o Library Solution (LIS), -si se requiere-, Flow Cell Tether (FCT) y un tubo de Flow Cell Flush (FCF) a temperatura ambiente. Agitar en vórtex. Centrifugar brevemente y poner en hielo.
Añadir seroalbúmina bovina (BSA) en una concentración total de 0,2 mg/ml a la mezcla de preparación de la celda de flujo, contribuye a que los resultados de la secuenciación sean óptimos y aumenta la producción de las celdas de flujo MinION R10.4.1 (FLO-MIN114).
Nota: no recomendamos utilizar ningún otro tipo de albúmina (p. ej., seroalbúmina humana recombinante).
Preparar la mezcla de acondicionamiento con seroalbúmina bovina, Flow Cell Flush (FCF) y Flow Cell Tether (FCT) como se indica a continuación. Mezclar con la pipeta a temperatura ambiente.
En un tubo proporcionado a la cantidad de celdas de flujo que se vayan a utilizar, mezclar los siguientes reactivos:
| Reactivo | Volumen por celda de flujo |
|---|---|
| Flow Cell Flush (FCF) | 1 170 µl |
| Bovine Serum Albumin (BSA) a una concentración de 50 mg/ml | 5 µl |
| Flow Cell Tether (FCT) | 30 µl |
| Volumen total | 1 205 µl |
Abrir la tapa del dispositivo MinION o GridION y deslizar la celda de flujo debajo del clip. Presionar la celda de flujo con firmeza para asegurar un contacto eléctrico y térmico adecuados.

Antes de cargar la biblioteca, verificar la celda de flujo para determinar el número de poros disponible.
Si se ha verificado la celda de flujo con anterioridad, este paso se puede omitir.
Dispone de más información en las instrucciones de comprobación de la celda de flujo del protocolo de MinKNOW.
Deslizar la tapa del puerto de purgado en el sentido de las agujas del reloj.

Tenga cuidado a la hora de extraer solución amortiguadora de la celda de flujo. No retire más de 20-30 μl y asegúrese de que la solución cubra la matriz de poros en todo momento. La introducción de burbujas de aire en la matriz puede dañar los poros de manera irreversible.
Tras abrir el puerto de purgado, comprobar si hay burbujas de aire bajo la tapa. Retirar una pequeña cantidad de solución amortiguadora para quitar las burbujas:
- Ajustar una pipeta P1000 a 200 μl.
- Introducir la punta de la pipeta en el puerto de purgado.
- Girar la rueda hasta que el indicador de volumen marque 220-230 μl o hasta que se pueda ver una pequeña cantidad de solución amortiguadora entrar en la punta de la pipeta.
Nota: Comprobar que haya un flujo continuo de solución amortiguadora circulando desde el puerto de purgado a través de la matriz de poros.

Cargar 800 μl de mezcla de acondicionamiento por del puerto de purgado, evitando introducir burbujas de aire. Esperar cinco minutos. Durante este tiempo, preparar la biblioteca para cargar siguiendo los pasos a continuación.

El vial Library Beads (LIB) contiene microesferas en suspensión. Las microesferas sedimentan muy rápido; por eso, es fundamental mezclarlas justo antes de usar.
En la mayoría de experimentos de secuenciación recomendamos utilizar Library Beads (LIB). El reactivo Library Solution (LIS) está disponible en caso de utilizar bibliotecas más viscosas.
Mezclar minuciosamente con la pipeta el contenido del vial Library Beads (LIB).
En un tubo nuevo de 1,5 ml Eppendorf DNA LoBind, preparar la biblioteca de la siguiente manera:
| Reactivo | Volumen por celda de flujo |
|---|---|
| Sequencing Buffer (SB) | 37,5 µl |
| Library Beads (LIB) mezcladas justo antes de usar, o Library Solution (LIS), si se requiere | 25,5 µl |
| Biblioteca de ADN | 12 µl |
| Total | 75 µl |
Terminar de acondicionar la celda de flujo:
- Levantar con suavidad la tapa del puerto de carga SpotON.
- Cargar 200 µl de mezcla de acondicionamiento en el puerto de purgado (no en el puerto SpotON), evitando introducir burbujas de aire.

Mezclar la biblioteca suavemente con la pipeta, justo antes de cargar.
Añadir, gota a gota, 75 μl de la biblioteca preparada en el puerto SpotON de la celda de flujo. Procurar que cada gota fluya hacia adentro del puerto antes de añadir la siguiente.

Volver a colocar con cuidado, la tapa del puerto SpotON, procurando que el tapón encaje en el agujero y cerrar el puerto de purgado.


Para obtener resultados de secuenciación óptimos, coloque la pantalla protectora sobre la celda de flujo justo después de cargar la biblioteca.
Recomendamos colocar la pantalla protectora en la celda de flujo y dejarla puesta mientras la biblioteca esté cargada, incluyendo los lavados y pasos de recarga. Retirar la pantalla cuando se haya extraído la biblioteca de la celda de flujo.
Colocar la pantalla protectora de la siguiente manera:
Colocar con cuidado el borde delantero de la pantalla protectora contra el clip.
Nota: no hacer fuerza sobre ella.Posar la pantalla protectora sobre la celda de flujo. La pieza debe asentarse alrededor de la tapa SpotON y cubrir por completo la sección superior de la celda de flujo.

La pantalla protectora no está fijada a la celda de flujo. Una vez colocada, es necesario manipular la celda de flujo con cuidado.
Cerrar la tapa del dispositivo y configurar un experimento de secuenciación en MinKNOW.
Al insertar una celda de flujo en el MinION Mk1D, la tapa queda apoyada sobre la celda y deja un pequeño espacio alrededor de los bordes. Esta separación es normal y no afecta el rendimiento del dispositivo.
En esta sección de Preguntas frecuentes hay más información sobre el tema.

5. Adquisición de datos e identificación de bases
Cómo empezar a secuenciar
Una vez la celda de flujo esté cargada, el experimento se pone en marcha desde MinKNOW, el programa de secuenciación que controla el dispositivo, la adquisición de datos y la identificación de bases en tiempo real. Encontrará intrucciones de uso más detalladas en el protocolo de MinKNOW.
Es posible utilizar y configurar MinKNOW para secuenciar de diferentes maneras:
- En un ordenador conectado a un dispositivo de secuenciación, ya sea directamente o a distancia.
- Directamente desde un dispositivo de secuenciación GridION, MinION Mk1C o PromethION 24/48.
Encontrará más información sobre el uso de MinKNOW en los manuales de usuario de los dispositivos:
Cómo empezar un experimento de secuenciación en MinKNOW:
1. Ir a la página principal y pulsar "Iniciar secuenciación".
2. Introducir los datos del experimento, como el nombre, la posición de la celda de flujo y el identificador de la muestra.
3. En la pestaña Kit, seleccionar Rapid Sequencing Kit (SQK-RAD114).
4. En la pestaña Configuración del experimento, ajustar los parámetros de secuenciación y salida del experimento o mantener la configuración por defecto.
Nota: si la identificación de bases estaba desactivada durante la configuración de un experimento, es posible identificar las bases tras la ejecución en MinKNOW. Encontrará más información en el protocolo de MinKNOW.
5. Al pulsar Inicio, se pondrá en marcha la secuenciación.
Análisis de datos
Una vez la secuenciación ha finalizado, es posible reutilizar o devolver la celda de flujo, como se describe en la sección sobre Reutilización o retorno de celdas de flujo.
Tras secuenciar e identificar las bases, es posible analizar los datos. Si desea más información sobre las opciones de análisis disponible durante y tras la identificación de bases, consulte el documento Data Analysis.
En la sección Análisis, se describen otras opciones para analizar los datos.
6. Reutilización y devolución de celdas de flujo
Material
- Kit Flow Cell Wash (EXP-WSH004)
Si al terminar el experimento desea volver a usar la celda de flujo, siga las instrucciones del protocolo Flow Cell Wash Kit y guarde la celda de flujo lavada a entre 2 °C y 8 ⁰C.
El protocolo Flow Cell Wash Kit está disponible en la comunidad Nanopore.
Una vez terminado el experimento, recomendamos lavar la celda de flujo cuanto antes. Si no es posible, se puede dejar en el dispositivo y lavar al día siguiente.
Otra posibilidad es seguir el procedimiento de devolución, lavar la celda de flujo y enviarla a Oxford Nanopore.
Aquí están las instrucciones para devolver celdas de flujo.
Ante cualquier duda o pregunta, consulte el apartado Resolución de problemas, que se encuentra en la versión en línea de este protocolo.
7. Análisis de datos
Análisis posterior a la identificación de bases
Existen varias opciones para completar el análisis de los datos de bases identificadas:
Flujos de trabajo de EPI2ME
A fin de realizar un análisis en profundidad de los datos, Oxford Nanopore Technologies ofrece una serie de tutoriales sobre bioinformática y flujos de trabajo, que están disponibles en EPI2ME. La plataforma proporciona un espacio donde los flujos de trabajo que depositan en GitHub nuestros equipos de Investigación y Aplicaciones, se pueden exponer con textos descriptivos, código bioinformático funcional y datos de ejemplo.
Herramientas de análisis orientadas a la investigación
Para realizar un análisis de datos más exhaustivo, Oxford Nanopore Technologies ofrece una serie de tutoriales y flujos de trabajo bioinformáticos, disponibles en EPI2ME Labs, que encontrará en la sección del mismo nombre de la comunidad Nanopore. La plataforma proporciona un espacio donde los proyectos que depositan en GitHub nuestros equipos de Investigación y Aplicaciones, se pueden exponer con textos descriptivos, código bioinformático funcional y datos de ejemplo.
Herramientas de análisis desarrolladas por la comunidad
Si no se proporciona en ninguno de los recursos anteriores un método de análisis que responda a las necesidades de investigación requeridas, consulte el centro de recursos Resource Centre y busque herramientas bioinformáticas para su aplicación. Varios miembros de la comunidad Nanopore han desarrollado sus propias herramientas y cartera de productos en desarrollo para analizar los datos de la secuenciación por nanoporos. La mayoría de ellas está disponible en GitHub. Oxford Nanopore Technologies no desarrolla ni mantiene esas herramientas y no garantiza que sean compatibles con la última configuración de química/programas informáticos.
8. Problemas durante la extracción de ADN y la preparación de bibliotecas
A continuación hay una lista de los problemas más frecuentes, con algunas posibles causas y soluciones propuestas.
También disponemos de una página de preguntas frecuentes, FAQ, en la sección Support de la comunidad Nanopore.
Si ha probado las soluciones propuestas y continúa teniendo problemas, póngase en contacto con el departamento de asistencia técnica, bien por correo electrónico (support@nanoporetech.com) o a través del chat Live Support de la comunidad Nanopore.
Baja calidad de la muestra
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Baja pureza del ADN (la lectura del Nanodrop para ADN OD 260/280 es <1,8 y OD 260/230 es <2,0-2,2) | El método de extracción de ADN no proporciona la pureza necesaria | Los efectos de los contaminantes se muestran en la página Contaminants. Probar con un método de extracción alternativo que no provoque el arrastre de contaminantes. Considere realizar un paso adicional de limpieza SPRI. |
Escasa recuperación de ADN tras la limpieza con microesferas magnéticas AMPure
| Observación | Posible causa | Comentarios y acciones recomendadas |
|---|---|---|
| Escasa recuperación | Pérdida de ADN debido a una proporción de microesferas magnéticas AMPure por muestra inferior a lo previsto. | 1. Las microesferas magnéticas AMPure sedimentan con rapidez; antes de añadirlas a la muestra hay que asegurarse de que estén bien resuspendidas. 2. Si la proporción de microesferas por muestra es inferior a 0.4:1, los fragmentos de ADN, sean del tamaño que sean, se perderán durante la limpieza. |
| Escasa recuperación | Los fragmentos de ADN son más cortos de lo esperado | Cuanto menor sea la proporción de microesferas magnéticas AMPure por muestra, más rigurosa será la selección de fragmentos largos frente a los cortos. Determinar siempre la longitud de la muestra de ADN en un gel de agarosa u otros métodos de electroforesis en gel, y, a continuación, calcular la cantidad adecuada de microesferas magnéticas que se debe utilizar. 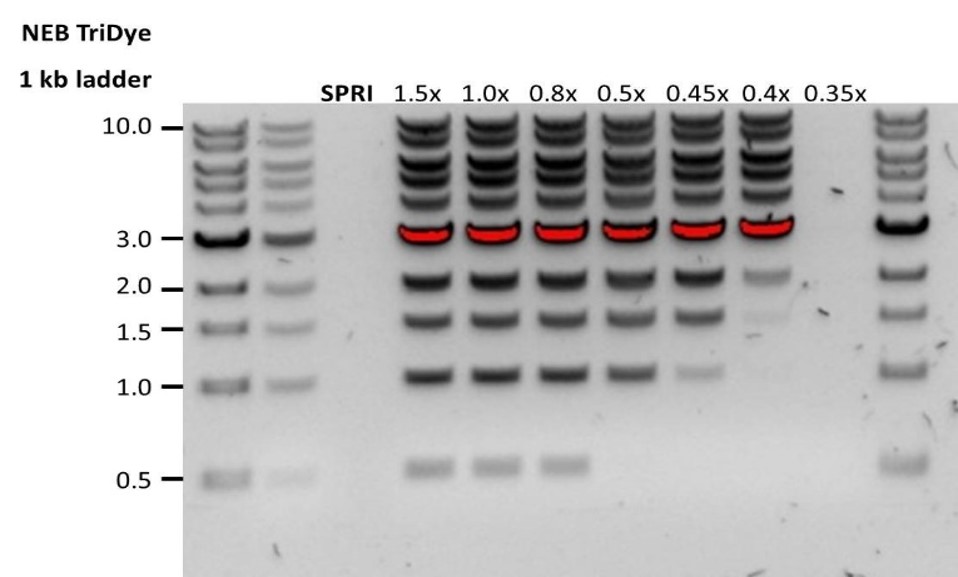 |
| Escasa recuperación tras la preparación de extremos | El paso de lavado utilizó etanol a <70 % | Cuando se utilice etanol a <70 %, el ADN se eluirá de las microesferas magnéticas. Asegúrese de utilizar el porcentaje correcto. |
9. Problemas durante el experimento al utilizar un kit de secuenciación de base rápida
A continuación hay una lista de los problemas más frecuentes, con algunas posibles causas y soluciones propuestas.
También disponemos de una página de preguntas frecuentes, FAQ, en la sección Support de la comunidad Nanopore.
Si ha probado las soluciones propuestas y continúa teniendo problemas, póngase en contacto con el departamento de asistencia técnica, bien por correo electrónico (support@nanoporetech.com) o a través del chat Live Support de la comunidad Nanopore.
Menos poros al inicio de la secuenciación que tras verificar la celda de flujo
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| MinKNOW presentó al inicio de la secuenciación un número de poros inferior al indicado durante la comprobación de la celda de flujo | Se introdujo una burbuja de aire en la matriz de nanoporos | Tras comprobar el número de poros presente en la celda de flujo, y antes de acondicionarla, es imprescindible quitar las burbujas que haya cerca del puerto de purgado. Si no se quitan, pueden desplazarse a la matriz de nanoporos y dañar de manera irreversible los nanoporos expuestos al aire. La forma más eficaz de evitar que esto ocurra se muestra en los vídeos Cómo cargar celdas de flujo MinION y Cómo cargar celdas de flujo PromethION. |
| MinKNOW presentó al inicio de la secuenciación un número de poros inferior al indicado durante la comprobación de la celda de flujo | La celda de flujo no está colocada correctamente | Detener el ciclo de secuenciación, quitar la celda de flujo del dispositivo e insertarla de nuevo. Comprobar que está firmemente asentada en el dispositivo y que ha alcanzado la temperatura deseada. Si procede, probar con una posición diferente del dispositivo (GriION/PromethION). |
| MinKNOW presentó al inicio de la secuenciación un número de poros inferior al indicado durante la comprobación de la celda de flujo | La presencia de contaminantes en la biblioteca podría haber dañado o bloqueado los poros | El número de poros resultante tras la evaluación de la celda de flujo se realiza usando el control de calidad de las moléculas de ADN presentes en el tampón de almacenamiento de la celda de flujo. Al inicio de la secuenciación, se utiliza la misma biblioteca para estimar el número de poros activos. Por este motivo, se estima que puede haber una variabilidad del 10 % en el número de poros detectados. Tener un número de poros considerablemente inferior al inicio de la secuenciación podría deberse a la presencia de contaminantes en la biblioteca que hayan dañado las membranas o bloqueado los poros. Para mejorar la pureza del material de entrada tal vez sea necesario usar métodos de purificación o extracción de ADN/ARN alternativos. Los efectos de los contaminantes están descritos en la página Contaminants. Se recomienda, probar con un método de extracción alternativo que no provoque el arrastre de contaminantes. |
Error en el script de MinKNOW
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| MinKNOW muestra el mensaje "Error en el script" | Reiniciar el ordenador y reiniciar MinKNOW. Si el problema continúa, reúna los archivos de registro MinKNOW log files y contacte con el servicio de asistencia técnica. Si no dispone de otro dispositivo de secuenciación, recomendamos que guarde la celda de flujo cargada a 4 °C y contacte con el servicio de asistencia técnica para recibir instrucciones de almacenamiento adicionales. |
Ocupación de poro por debajo del 40 %
| Observación | Posible causa | Comentarios y acciones recomendadas |
|---|---|---|
| Ocupación de poro <40 % | No se cargó suficiente cantidad de biblioteca en la celda de flujo | Procurar cargar la concentración correcta de una biblioteca de buena calidad en una celda de flujo MinION o GridION. Para comprobar la concentración requerida, consultar la sección Preparación de la biblioteca del protocolo. Cuantificar la biblioteca antes de cargarla y calcular moles con herramientas como la calculadora Biomath de Promega, (opción "dsDNA: μg to pmol"). |
| Ocupación de poro próxima a 0 | Se utilizó el kit Rapid Sequencing Kit V14 o Rapid Barcoding Kit V14 y los adaptadores de secuenciación no se ligaron al ADN | Seguir el protocolo paso a paso y utilizar los volúmenes y las temperaturas de incubación correctos. También se puede preparar una biblioteca de control con lambda para valorar la integridad de los reactivos. |
| Pore occupancy close to 0 | No hay anclaje (tether) en la celda de flujo | Los anclajes se añaden durante el cebado de la celda de flujo (vial FCT). No olvide añadir el anclaje (vial FCT) al tubo de enjuague de la celda de flujo (vial FCF) antes del acondicionamiento. |
Longitud de lectura más corta de lo esperado
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Longitud de lectura más corta de lo esperado | Fragmentación no deseada de la muestra de ADN | La longitud de lectura refleja la longitud del fragmento de la muestra de ADN. La muestra de ADN se puede fragmentar durante la extracción y preparación de la biblioteca. 1. Consulte la sección de buenas prácticas de extracción en la página Extraction Methods de la comunidad Nanopore. 2. Visualizar la distribución de la longitud de los fragmentos de las muestras de ADN en un gel de agarosa antes de proceder a la preparación de la biblioteca. 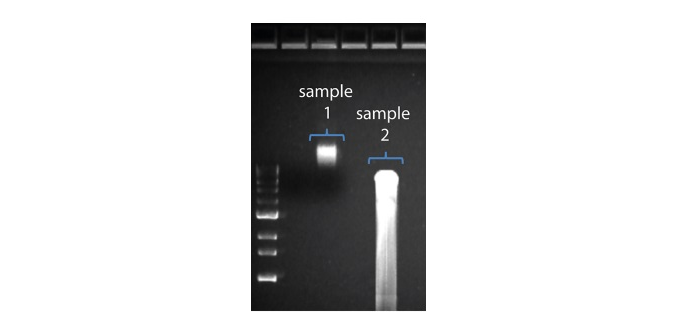 En la imagen superior, la muestra 1 contiene alto peso molecular, mientras que la muestra 2 se ha fragmentado. En la imagen superior, la muestra 1 contiene alto peso molecular, mientras que la muestra 2 se ha fragmentado.3. Durante la preparación de la biblioteca, evitar pipetear y agitar en vórtex cuando se mezclen los reactivos. Dar suaves golpes con el dedo o invertir el vial es suficiente. |
Gran proporción de poros no disponibles
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
Gran proporción de poros no disponibles (se muestran en azul en el panel de canales y en el gráfico de actividad de poros) 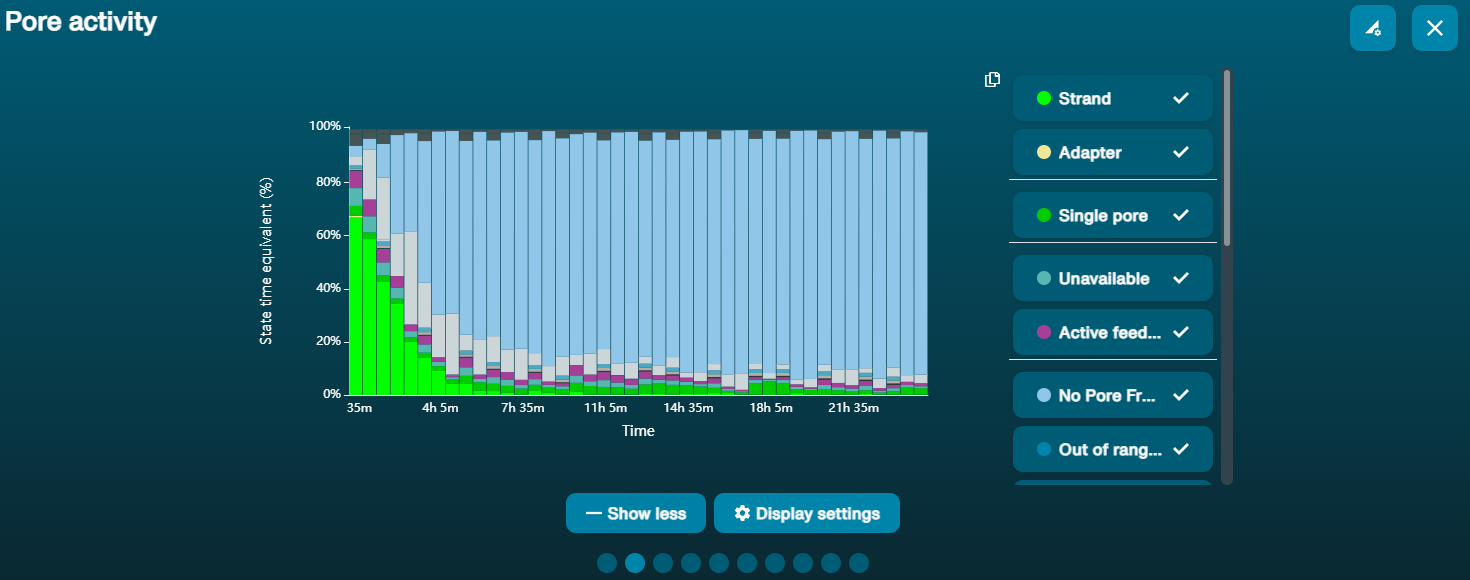 Conforme pasa el tiempo, el gráfico de actividad de poros de arriba muestra una proporción creciente de poros "no disponibles". Conforme pasa el tiempo, el gráfico de actividad de poros de arriba muestra una proporción creciente de poros "no disponibles". | Hay contaminantes presentes en la muestra | Algunos contaminantes se pueden eliminar de los poros mediante la función de desbloqueo incorporada en MinKNOW. Si funciona, el estado de los poros cambiará a "sequencing pores". Si la porción poros no disponibles se mantiene elevada o aumenta: 1. Realizar un purgado de nucleasa con el kit de lavado Flow Cell Wash Kit (EXP-WSH004) 2. Realizar varios ciclos de PCR para intentar diluir cualquier contaminante que pueda estar causando problemas. |
Gran proporción de poros inactivos
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Gran proporción de poros inactivos/no disponibles (se muestran en azul claro en el panel de canales y en el gráfico de actividad de poros. Los poros o membranas están dañados de manera irreversible) | Se han introducido burbujas de aire en la celda de flujo | Las burbujas de aire introducidas durante el acondicionamiento de la celda y carga de la biblioteca podrían dañar los poros de forma permanente. La forma más eficaz de evitar que esto ocurra se muestra en los vídeos Cómo cargar celdas de flujo MinION y Cómo cargar celdas de flujo PromethION. |
| Gran proporción de poros inactivos/no disponibles | Ciertos compuestos copurificados con ADN | Compuestos conocidos, incluidos los polisacáridos, se asocian generalmente con el ADN genómico de las plantas. 1. Consulte los métodos de extracción de ADN en la página Plant leaf DNA extraction method. 2. Purificar con el kit QIAGEN PowerClean Pro. 3. Realizar una amplificación del genoma completo con la muestra original de ADNg utilizando el kit QIAGEN REPLI-g. |
| Gran proporción de poros inactivos/no disponibles | Hay contaminantes presentes en la muestra | Los efectos de los contaminantes se muestran en la página Contaminants. Probar con un método de extracción alternativo que no provoque el arrastre de contaminantes. |
Fluctuación de la temperatura
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| Fluctuación de la temperatura | La celda de flujo ha perdido contacto con el dispositivo | Comprobar que una almohadilla térmica cubra la placa metálica de la parte posterior de la celda de flujo. Reinsertar la celda de flujo y presionar para asegurarse de que las clavijas del conector estén bien conectadas al dispositivo. Si el problema continúa, contacte con el servicio de asistencia técnica. |
Error al intentar alcanzar la temperatura deseada
| Observaciones | Posibles causas | Comentarios y acciones recomendadas |
|---|---|---|
| MinKNOW muestra el mensaje "Error al intentar alcanzar la temperatura deseada" | El dispositivo ha sido colocado en un lugar con una temperatura ambiente inferior a la media o en un lugar con escasa ventilación (lo que provoca el sobrecalientamiento de las celdas de flujo). | MinKNOW dispone de un tiempo predeterminado para que las celdas de flujo alcancen la temperatura fijada. Una vez transcurrido ese tiempo, aparece un mensaje de error, pero el experimento de secuenciación continúa. Secuenciar a una temperatura incorrecta puede provocar disminuciones en el rendimiento y generar índices de calidad Qscore menores. Corrija la ubicación del dispositivo, procurando que esté a temperatura ambiente y tenga buena ventilación; a continuación, reinicie el proceso en MinKNOW. Encontrará más información sobre el control de temperatura del MinION en este enlace. |







